|
VERSION 2013 en travaux retour panorama, plan du cours de 1èreS |
|
à la théorie de l'information génétique |
||||||||||||||||||||||||||||
|
Plan
TD10 - Simualisation de molécules avec Jmol (rappels ADN et ARN), aa, protéines TD11 - La matière du vivant TD12 - Réplication-hybridation et PCR TD13 - synthèse des protéines TD14 - Génomes TD15 - Gènes héréditaires TD16 - Maladies géniques |
|
FIN DE LA PARTIE STRICTEMENT SCOLAIRE DÉBUT DE LA PARTIE intellectuellement stimulante
|
||||||||||||||||||||||||||||
|
Une grande ressemblance : acides nucléiques et peptides 1 - Les acides nucléiques et les protéines sont des polymères |
|
|
||||||||||||||||||||||||||||
| |
|
1.1 - Les acides nucléiques sont de longs polymères de nucléotides |
||||||||||||||||||||||||||||
| |
Les acides nucléiques sont des polymères (du grec poly = plusieurs et merein = partage) (voir cours de seconde) car il sont composés d'unités identiques : les monomères. Quand il y a plusieurs types de monomères on parle de copolymères. Les nucléotides sont des molécules composées d'une base azotée (A= adénine, T=thymine, U=uracile, C=cytosine, G=guanine), d'un sucre (ribose ou désoxyribose) et d'un, deux ou trois groupement(s) phosphate(s). |
|
Les nucléotides ne sont pas que des composants des acides nucléiques mais ont des fonctions variées dans la cellule: par exemple l'ATP (adénosine triphosphate = adénine + ribose (l'adénosine est le nom du nucléoside) + 3 phosphates) et ses dérivés monophosphate (AMP) ou diphosphate (ADP) qui sont des molécules énergétiques intermédiaires (transfert de l'énergie chimique de liaison entre molécules) mais aussi informatives (AMP cyclique ou cAMP par exemple); ou le dGTP (adénine+désoxyribose+3 groupements phosphate; le "d" devant la molécule indiquant la nature du sucre: le désoxyribose) qui est aussi une molécule énergétique. |
|||||||||||||||||||||||||||
| |
|
1.1.1 - L'ADN est une longue molécule en hélice à deux brins |
||||||||||||||||||||||||||||
| |
L'ADN (acide désoxyribonucléique)
est formé de deux chaînes (brins) de
nucléotides dessinant une double hélice
dont le diamètre est de 2 nm. |
|
- Les nucléotides d'une chaîne sont
associés entre eux par des liaisons covalentes (entre
le groupement phosphate d'un nucléotide et le
désoxyribose d'un autre nucléotide). |
|||||||||||||||||||||||||||
|
En 1èreS il est nécessaire de comprendre, en plus de ce que vous avez vu en seconde, l'orientation (antiparallèle) des deux brins (5'-> 3' et 3'->5') 3' et 5' désignent les numéros des carbone du désoxyribose. |
|
|
||||||||||||||||||||||||||||
| |
|
|
|
1.1.2 - Les ARN sont des chaînes à un seul brin |
||||||||||||||||||||||||||
| |
|
Les ARN (acides ribonucléiques)
sont des chaînes (un seul brin) de nucléotides
pouvant se replier et leurs bases s'apparier sur
de courtes distances. |
||||||||||||||||||||||||||||
| |
|
1.2 - Les protéines sont de longs polymères d'acides aminés |
||||||||||||||||||||||||||||
|
Les acides aminés et les protéines |
|
Les chaînes d'aa (aa
réunis par des liaisons peptidiques)
sont des peptides . De 2 à 10 aa on
parle d'oligopeptides; au-delà on
parle de polypeptides. Le terme protéine
(s.s) est réservé aux polyppeptides
dont le poids moléculaire est
supérieur à 10.000 daltons (un
dalton est le poids d'un atome d'hydrogène).
En pratique, il n'est pas rare d'utiliser
abusivement le mot protéine comme
équivalent de peptide. |
|
Il existe 20 acides aminés (aa) dans les
protéines du vivant. |
|
Les protéines sont souvent formées de l'assemblage de plusieurs molécules et contiennent des éléments non peptidiques (appelés groupement prosthétique, comme le groupe tétrapyrolique à Fe qui forme l'hème et s'ajoute à la molécule de globine de l'hémoglobine... elle-même formée de 4 sous-unités identiques 2 à 2).
|
||||||||||||||||||||||||
|
Remarques importantes : |
|
La suite linéaire (en ligne) des nucléotides d'une chaîne de l'ADN (l'autre étant déterminée par complémentarité des bases), ou de l'unique chaîne des ARN, forme une séquence propre à chaque molécule d'acide nucléique. Elle peut être représentée par la suite des bases (par exemple .....AACTAGGCTTAA....). De même la suite linéaire des aa d'une protéine est appelée séquence ou structure primaire. |
|
Les séquences constituent une information linéaire qui n'est pas sans rappeler l'information exprimée par la suite de lettres d'un mot. Mais ce n'est pas la seule information que peut donner une molécule qui se déploie dans l'espace avec une forme. L'idée selon laquelle la séquence est la cause de la forme de la molécule est suivie pour les peptides et partiellement pour les ARN mais pas pour l'ADN. Pourquoi ?  Même si la forme est indubitablement liée à la séquence elle n'en est pas la résultante car la forme dépend aussi des conditions de milieu et de molécules additionnelles pouvant s'ajouter pour donner une structure complexe. Par exemple l'ADN s'associe avec des protéines pour donner une fibre chromatinienne (et un chromosome) chez les eucaryotes et les protéines sont nombreuses à s'associer entre elles ou avec des ARN pour donner des complexes. Le nombre de formes (motifs) est relativement limité (domaines) mais les associations sont très nombreuses. |
||||||||||||||||||||||||||
|
D'où vient l'ADN ? 2 - L'ADN est soit hérité soit synthétisé à partir d'un autre acide nucléique |
||||||||||||||||||||||||||||||
| |
|
2.1 - La division cellulaire répartit l'ADN dans les deux cellules filles |
||||||||||||||||||||||||||||
|
L'ADN est transmis par hérédité d'une cellule à l'autre lors de la division |
|
Chez les Procaryotes, l'ADN synthétisé
pendant la phase de croissance est réparti (mais
pas forcément équitablement, cela dépend
du nombre de molécules d'ADN présentes) dans
les deux cellules filles lors de la DIVISION. Des échanges d'ADN sont très
fréquents entre bactéries d'une même
espèce par CONJUGAISON grâce à des ponts
cytoplasmiques établis au niveau des pili sexuels :
c'est la recombinaison bactérienne. Les
bactéries échangeuses sont apellées
conjugantes. |
|
Chez les eucaryotes la mitose répartit de
façon égale (sauf anomalie) par clivage des
chromosomes, l'ADN synthétisé pendant la phase S
de l'interphase et condensé au niveau des chromosomes.
|
|
En terminale nous verrons un mécanisme autre de division qui se rattache à la reproduction sexuée : la méïose. L'héritage y est fortement inégal. Chaque gamète n'a que la moitié de l'ADN de la cellule mèrel (c'est la réduction chromatique). Les ADN des gamètes sont réunis lors de la FECONDATION. Mais l'ADN n'est pas le seul acide nucléique hérité : chez certains amphibiens, les ARN ovocytaires sont synthétisés en grande quantité et servent dans les premières phases de développement embryonnaire. Le developpement se fait donc surtout à partir d'ARN maternels avant que de nouveaux ARN embryonnaires soient transcrits. |
||||||||||||||||||||||||
| |
|
2.2 - REPLICATION - Avant la division, de grandes molécules d'ADN sont répliquées par un complexe enzymatique : l'ADN polymérase |
||||||||||||||||||||||||||||
|
Electronographies de fibres chromatinienne en duplication.... .... revoir les différents états de l'ADN au cours du cycle cellulaire
PAGE AVEC VERSIONS FRANÇAISES des vidéos |
|
La réplication
de l'ADN est une synthèse de
deux molécules d'ADN identiques à partir d'une
molécule d'ADN. Chaque molécule d'ADN
fille est composée d'un brin ancien et d'un brin
nouveau complémentaire du brin ancien; on dit que la
réplication est semi-conservative. La
réplication de l'ADN est catalysée par un gros
complexe enzymatique contenant plus de 20 enzymes (chez
les procaryotes) mais
surtout l'ADN polymérase (les
complexes sont moins bien connus chez les eucaryotes). |
|
L'ADN polymérase nécessite une molécule d'ADN à répliquer, des désoxyribonucléotides, de l'énergie (ATP) et un petit fragment d'ARN complémentaire d'une petite séquence de l'ADN à répliquer (amorce). Il existe plusieurs ADN polymérases chez les bactéries (au moins 3) et plus encore chez les eucaryotes. |
||||||||||||||||||||||||||
| |
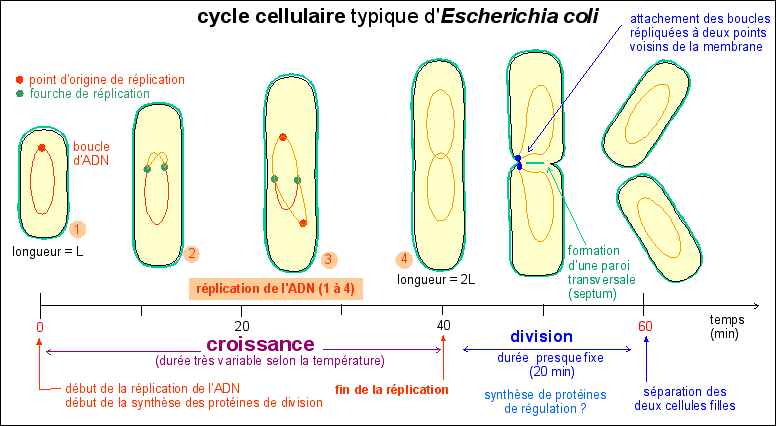
L'ADN d'une cellule est répliqué EN ENTIER au cours de la phase de croissance du cycle cellulaire. La réplication se déroule avec une fidélité excellente: chez E. coli on estime le nombre d'erreurs de copie à 1 pour 109 à 1010 nucléotides, ce qui fait, pour un génome évalué à 4,7.106 paires de bases, une erreur pour 1.000 à 10.000 réplications. Il existe des systèmes de réparation de l'ADN qui utilisent aussi une ADN polymérase. La vitesse de polymérisation (ajout des désoxyribonucléotides pour former le nouveau brin) est de l'ordre de 200 à 1.000 nucléotides par seconde chez E. coli. |
|
Le problème du déterminisme du cycle cellulaire est aussi celui du lien entre la croissance cellulaire et la réplication de l'ADN.... il est loin d'être résolu. En tout cas il ne sera pas traité dans ces pages. |
|||||||||||||||||||||||||||
|
Analyse
de l'article scientifique relatant l'expérience: |
|
Expérience de Meselson et Stahl (1958) présentation scolaire.... |
||||||||||||||||||||||||||||
| |
|
Escherichia coli est cultivée pendant de nombreuses générations (= durée d'un cycle cellulaire puisqu'une génération sépare une cellule mère de sa cellule fille) sur un milieu de culture contenant des désoxyribonucléotides marqués à l'15N (témoin 1). Cette souche de départ est transférée pendant un génération sur un milieu contenant uniquement des désoxyribonucléotides marqués à l'14N (expérience 1). Enfin on transfère des bactéries issues de cette culture à nouveau sur un milieu contenant uniquement des désoxyribonucléotides marqués à l'15N (expérience 2). |
|
La mesure de la quantité de désoxyribonucléotides incorporés dans l'ADN lors de la phase de réplication (croissance cellulaire) se fait à l'aide de prélèvement de bactéries dont l'ADN est extrait et centrifugé sur gradient de densité; l'ADN se plaçant plus ou moins bas dans le tube selon sa densité. La bande d'ADN, fluorescente sous U.V. est révélée par éclairement U.V. du tube. Une densité de 1,8 signifiant un ADN ne contenant quasiment que de l'15N et une densité de 1,65 signifiant un ADN ne contenant quasiment que de l'14N. |
|
Le tube 2 correspond à un témoin d'une souche cultivée pendant de nombreuses génération sur sur milieu contenant des désoxyribonucléotides marqués à l'14N. Le tube 5 correspond à une génération supplémentaire sur milieu contenant des désoxyribonucléotides marqués à l'14N (expérience 3). |
||||||||||||||||||||||||
|
|
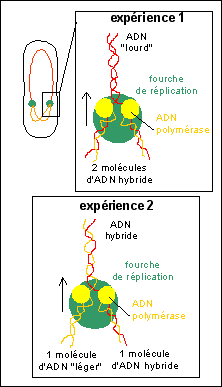 Une illustration de
l'interprétation du mécanisme de
réplication semi-conservative de l'ADN dans
l'expérience de Meselson et Stahl.
Une illustration de
l'interprétation du mécanisme de
réplication semi-conservative de l'ADN dans
l'expérience de Meselson et Stahl. |
||||||||||||||||||||||||||||
|
interprétation: Les témoins correspondent à des bactéries dont la quasi-totalité de l'ADN est composé de désoxyribonucléotides possédant de l'14N (témoin 2) qualifié d'ADN "léger" ou de l'15N (témoin 1) qualifié d'ADN "lourd". |
|
Dans l'expérience 1 (tube 3) la position de l'ADN intermédiaire en densité entre l'ADN léger et de l'ADN lourd permet de dire que le mécanisme de réplication de l'ADN, qui s'est normalement déroulé une seule fois depuis le transfert sur milieu léger, est semi-conservatif. En effet, les molécules d'ADN obtenues lors de la réplication contiennent TOUTES pour moitié de l'14N et de l'15N. Ce qui peut s'expliquer aisément si l'on imagine un système de réplication qui conserve un des brins anciens dans chaque nouvelle molécule et synthétise un brin nouveau complémentaire. Les deux molécules d'ADN issues d'une telle réplication sont donc identiques et pour un brin (nouveau) composées d'14N et pour l'autre brin (ancien) composées d'15N. On peut parler de molécules hybrides 14N-15N. |
|
Lors d'une seconde réplication avec des désoxyribonucléotides contenant de l'14N (milieu "léger"), les molécules hybrides sont aussi répliquées selon un mécanisme semi-conservatif qui donnera une molécule d'ADN léger et une molécule d'ADN hybride à partir de chaque molécule ancienne (hybride). Une troisième réplication sur "milieu léger" donnera aussi ces deux types de molécules mais dans un rapport différent. Il y aura une molécule hybride pour 3 molécules d'ADN léger. |
|
|||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||
|
Analyse
de l'article scientifique relatant la véritable
expérience de Taylor : |
|
Expérience de Taylor (1957) présentation scolaire modifiée |
||||||||||||||||||||||||||||
| |
|
On utilise des plantules de Vicia faba ( qui possède 12 chromosomes) dont les racines sont plongées dans des solutions contenant ou non un nucléoside (base + sucre) marqué radioactivement : la thymidine (thymine + désoxyribose) marquée par le tritium (3H ou 3T), isotope lourd et radioactif de l'hydrogène. Pour être incorporé à l'ADN en phase de synthèse, la thymidine doit être transformée en nucléotide par ajout d'un groupement phophate (en fait trois groupements puisque ce sont des nucléotides tri-phosphate qui sont utilisés lors de la réplication). |
|
L'incorporation du désoxyribonucléotide radioactif est suivi par des autoradiographies qui sont des expositions de préparations cellulaires à une plaque ou un film photographique. Les composés argentés de l'émulsion photographique précipitent lorsqu'ils rencontrent un électron émis par radioactivité ß- par les atomes de 3H incorporés dans le thymidine radioactive. Les grains d'argent forment des points noirs que le développement révèle. L'emplacement des points noirs indique donc de façon approximative la position des molécules de thymidine tritiée de la préparation cellulaire. Les temps d'exposition des émulsions photographiques sont bien sûr beaucoup plus longs que pour une impression par les photons. |
||||||||||||||||||||||||||
| |
|
Les racines de Vicia faba, sont cultivées pendant la durée d'un cycle cellulaire sur un milieu "chaud", c'est-à-dire contenant de la thymidine tritiée. Puis la division est bloquée en métaphase par l'utilisation de la colchicine (un extrait de la Colchique) qui empêche la cytodiérèse et la séparation des chromatides mais sans bloquer la condensation des chromosomes. Les cellules en métaphase de mitose sont repérées et certaines sont autoradiographiées. L'aspect d'un chromosome est représenté ci-dessous à gauche. Certaines de ces cellules sont replacées immédiatement sur un milieu "froid", c'est-à-dire dépourvu de thymidine radioactive, pendant la durée d'un deuxième cycle cellulaire. L'aspect d'un chromosome d'une autoradiographie d'une cellule en prophase de mitose est présentée ci-dessous à droite. |
|
|
|
1a, 1b : un chromosome à deux chromatides marquées d'une cellule à 12 chromosomes après une exposition de 10h à la colchicine. Les chromatides sont interprétées comme étant encore partiellement accolées. 2a, 2b : plusieurs chromosomes d'une cellule à 24 chromosomes après 34h d'exposition à la colchicine; pour l'interprétation de la différence entre chromatides et chromosomes (voir plus bas) on s'appuie sur l'interprétation liée à l'ADN (ce qui est un raisonnement erroné): la forme en "étoile à 3 branches" en haut à gauche ne peut, du fait de l'hypothèse interprétative d'un marquage sur une seule chromatide, que correspondre à deux chromosomes partiellement accolés; le chromosome le plus à droite, celui du centre et celui le plus en bas, seraient des chromosomes avec deux chromatides assez bien séparées et l'une des chromatides seulement marquée; le chromosome en L ouvert correspondrait à deux chromatides d'un même chromosome largement éloignées mais réunies au sommet... |
||||||||||||||||||||||||
| |
|
|
||||||||||||||||||||||||||||
|
Illustration de l'interprétation de l'expérience de Taylor (en orange: le centromère (qui n'est pas visible); les points noirs correspondent aux grains d'argent de l'autoradiographie) (schémas d'après Principe de Biochimie, Lehninger et al, 1994, Flammarion-Médecine-Sciences) |
|
|
||||||||||||||||||||||||||||
|
Explication de l'expérience: |
|
Tout en n'oubliant pas que le chromosome est une structure complexe de compaction de l'ADN nucléaire (comme le représente le schéma ci-contre - l'ADN d'une cellule humaine est estimé à environ 2 m soit quelques centimètres par chromosome pour un diamètre de 2 nm soit un rapport longueur sur diamètre de quelques dizaines de millions !!!!) on s'intéresse à la composition de chaque chromatide.
|
|
Les chromatides radioactives sont composées d'une molécule d'ADN hybride. C'est-à-dire que la molécule d'ADN hybride à incorporé dans UN de ses brins, le brin nouvellement formé, de la thymidine tritiée. Cette incorporation n'a lieu que pendant la phase S du cycle cellulaire (synthèse de l'ADN) et ne touche donc que les cellules qui étaient dans cette phase de l'interphase peu après le début de la mise en culture. Lorsque les cellules sont retirées du milieu chaud et observées, seules celles qui se trouvent maintenant en métaphase peuvent présenter de tels chromosomes. |
|
Le fait que les deux chromatides, issues de la réplication de l'ADN en phase S de l'interphase, sont TOUTES LES DEUX hybrides (c'est-à-dire composées d'un brin ancien, non radioactif et d'un brin nouveau ayant incorporé de la thymidine tritiée radioactive) peut être expliqué par un mécanisme semi-conservatif de la réplication de l'ADN, comme chez les Procaryotes. |
||||||||||||||||||||||||
|
|
|
2.3 - De petites molécules d'ADN peuvent être synthétisées à partir d'ARN |
||||||||||||||||||||||||||||
|
cours d'immunologie de TS sur le VIH (qui contient des molécules de transcriptase inverse comme tous les rétrovirus à ARN) |
|
L'ADN n'est pas uniquement synthétisé à partir d'ADN par réplication. Des molécules d'ADN courtes peuvent être synthétisées par une enzyme de type ADN polymérase mais qui travaille à partir d'un brin d'ARN et non d'un brin d'ADN. C'est l'ADNpolymérase ARNdépendante (ou transcriptase inverse). La synthèse est aussi apellée transcription inverse (voir ci-dessous). |
|
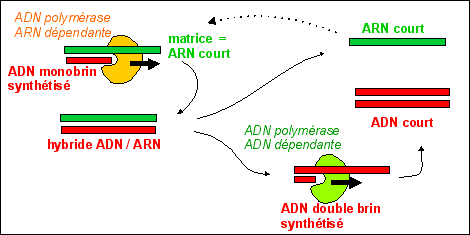 |
||||||||||||||||||||||||||
| |
|
Depuis les années 80-90, où on les recherche, on a trouvé des transcriptases inverses chez des bactéries, des mycètes et même chez les ovocytes de poissons. Mais pas dans toutes les cellules et on est loin de connaître l'importance de ce mécanisme. En l'état actuel des connaissances, on suppose qu'il est de bien moindre importance que la réplication. Ce mécanisme, s'il s'avérait être répandu changerait profondémment notre compréhension de l'information génétique (voir ci-dessous). |
|
En résumé: |
||||||||||||||||||||||||||
|
Les données certaines 3 - Les rôles indiscutables de l'ADN dans la synthèse des ARN et des protéines |
||||||||||||||||||||||||||
| |
|
3.1 - TRANSCRIPTION - L'ADN est transcrit en ARN par un complexe enzymatique : l'ARN polymérase |
||||||||||||||||||||||||
|
3.1.1 - La transcription est la copie de l'un des brins de l'ADN en ARN. |
|
|
||||||||||||||||||||||||
|
La transcription est la synthèse d'une molécule d'ARN (monobrin) à partir d'un l'un des brins d'une molécule d'ADN (brin matrice servant à une copie complémentaire base à base). Les deux brins peuvent chacun être transcrits et ne portent donc pas la même information génétique. (voir page sur les génomes.
PAGE AVEC VERSIONS FRANÇAISES des vidéos |
|
Lors de la transcription, la molécule d'ADN est maintenue ouverte (brins séparés) par un gros complexe enzymatique : l'ARN polymérase (ARN polymérase ADN dépendante qui contient du Zn2+) dans une zone d'une longueur de 17 paires de bases (3,4 nm pour 10 pb) ce qui nécessite, du fait de l'enroulement de l'ADN, de dérouler l'ADN en amont et de le réenrouler en aval de la zone de transcription. L'ARN polymérase copie un des brins de l'ADN (brin non codant ou brin ADN matrice ou brin transcrit) en un ARN complémentaire à une vitesse qui atteint 50 nucléotides à la seconde chez E. coli. La transcription nécessite l'ion Mg2+ et de l'énergie fournie par les ribonucléosides triphosphate (ATP, GTP, CTP et UTP). Toutes les étapes de la transcription sont régulées par des enzymes ou des facteurs par de nombreux mécanismes qui constituent le cœur de la régulation de l'expression génétique. La régulation consiste d'abord à déterminer le point de départ et le point d'arrêt de la transcription. La transcription a lieu dans le noyau interphasique, même pendant la phase S et est particulièrement intense chez les cellules qui réalisent des synthèses (cellules en croissance, cellules accumulant des réserves...). On connaît des cellules (ovocytes notamment bloqués au milieu de leur division) pour lesquelles la trancription se déroule aussi pendant la division cellulaire (chomosomes en écouvillons des amphibiens). Remarques: |
|
|
||||||||||||||||||||||
|
3.1.2 - Il existe plusieurs types d'ARN et la plupart subissent une maturation |
|
|
||||||||||||||||||||||||
|
Les transcrits primaires sont de longues molécules d'ARN qui donnent plusieurs types d'ARN différents :
voir la forme d'un ARNt sur la page générale de simualisation des molécules |
|
Les ARN qui restent dans le noyau sont : Les ARN qui migrent dans le cytoplasme par des pores
nucléaires vont participer à la synthèse
des protéines : Chez les eucaryotes, les transcrits primaires avant de devenir pré-ARN sont modifiés par l'ajout d'une coiffe (à l'extrémité 5') et d'une queue (à l'extrémité 3'). Chez les procaryotes, seule l'extrémité 5' possède des groupements phosphates supplémentaires (voir complément). |
|
Récemment, on a mis en évidence chez la
souris que les transcrits primaires sont constitués
d'une majorité d'immenses molécules dont
très peu contiennent des séquences correspondant
à des ARNm (voir Qu'est-ce
qu'un gène, p 3 réf 3). |
||||||||||||||||||||||
| |
|
L'épissage Chez les eucaryotes les ARNm proviennent de la maturation de longs pré-ARNm ; certaines portions de l'ARN prémessager -les introns- sont excisées, alors que certaines portions -les exons- sont épissés (l'épissage consiste en la ligature des exons). |
|
Le gène de la dystophine (DND)
est le plus long du génome humain (0,08%)
et contient de nombreux introns (79) . Il mesure
2400kb alors que l'ARNm est de 14kb (pour une
protéine de 3685 aa), Fichiers pdb: 1EG3,
1EG4,
1DXX).
À raison de 40 pb par seconde la
transcription du pré-ARNm dure près
de 17h. |
||||||||||||||||||||||
|
|
|
Pour la plupart des préARN l'épissage est réalisé au sein d'un splicéosome (complexe de 150 protéines et 5 ARN - petits ARN nucléaires : snRNA: small nuclear RNA), d'une taille semblable à celle du ribosome. Quelques introns sont auto-épissant (ils catalysent leur propre excision, ne pas oublier que ce sont des ARN, ceux qui ont une activité enzymatique sont apellés ribozymes). |
||||||||||||||||||||||||
|
L'épissage est un mécanisme très complexe qui peut encore nous réserver des surprises: |
|
- L'épissage est parfois alternatif (non
continu : il saute des exons) : c'est-à-dire que les
exons liés entre eux ne sont pas toujours les
mêmes (des introns peuvent même faire partie de
l'ARNm). On peut donc obtenir plusieurs types d'ARNm (donnant
des polypeptides alternatifs appelés isoformes)
à partir d'un seul long pré-ARNm. On estime que
75% des gènes humains (et 40% de ceux de la Drosophile)
subissent un épissage alternatif produisant plus d'une
isoforme. - On connaît des épissages entre des exons de deux ARNm différents (épissage trans). - Les pré-ARNm obtenus à partir d'un gène peuvent aussi être différents par modification du site de départ de la transcription. - L'épissage est régulé par des protéines qui en se fixant sur le pré-ARNm activent ou répriment l'épissage. De plus, on peut même obtenir plusieurs ARNm à partir d'un seul ARNprémessager. Chaque ARNprémessager ne correspond donc pas à un seul et unique ARNm. Bien que les termes d'exons (séquence
épissée) et d'intron (séquence
excisée) fassent référence à de
l'ARN, ils sont aussi employés pour désigner des
séquences d'ADN. |
|
La maturation des ARNm, étant donné sa complexité, est tout aussi importante dans l'expression de l'information génétique que celle de la transcription. Elle a lieu dans le noyau (seuls les ARN matures sont transportés dans le cytoplasme) : Remarque: |
||||||||||||||||||||||
| |
|
. Remarques : - L'ADN des mitochondries et des chloroplastes est transcrit directement au sein de ces organites. - L'ensemble des ARNm d'une cellule forme le transcriptome; pour un fibroblaste humain par exemple il peut comprendre jusqu'à 300.000 molécules d'ARNm (une cellule humaine comprendrait 21.000 gènes moléculaires environ dont plus de 50% seraient actifs mais avec des taux très variables selon le type de cellule, voir ENCODE), chaque ARNm n'étant présent que sous la forme d'au maximum 15 copies. On sait détecter par la technologie des puces à ADN (DNA chips) une unique copie d'un ARNm dans un broyat cellulaire... |
||||||||||||||||||||||||
| |
|
3.2 - TRADUCTION - Les ARNm sont traduits en polypeptides au sein des ribosomes dans le cytoplasme 80% de l'énergie cellulaire d'une bactérie à croissance rapide est consacrée à la synthèse des protéines. 50% du poids sec d'une telle bactérie peuvent constituer la machinerie de synthèse à partir des ARNm |
||||||||||||||||||||||||
|
3.2.1 - La traduction est une réaction enzymatique |
|
|
||||||||||||||||||||||||
|
Une enzyme est un catalyseur biologique (biocatalyseur); il accélère une réaction chimique (métabolique) en y participant et est restitué intègre en fin de réaction (voir ancienne page cours 1èreS). Les enzymes nécessitent des co-facteurs (éléments supplémentaires activant l'enzyme). Une réaction enzymatique est une réaction chimique (du métabolisme) qui est catalysée par des enzymes, ce qui peut s'appliquer à la plupart des réactions chimiques du vivant. |
|
La phase de traduction de la synthèse d'une
protéine nécessite: |
|
PAGE AVEC VERSIONS FRANÇAISES des vidéos |
||||||||||||||||||||||
|
|
|
Les ribosomes sont de gros complexes ARN-protéines composés de 2 sous-unités qui s'assemblent lors de la traduction (20 nm de ø environ, un peu plus petits chez les procaryotes, un peu plus gros chez les eucaryotes, MM>2,5MDa). Les ribosomes font partie des ribozymes (= ARN ayant des propriétés enzymatiques); la grande sous-unité, débarassée de la plupart de ses protéines est encore capable de catalyser la formation de liaisons peptidiques. Il n'est pas courant de dire que les ARNm et les ARNt qui participent à la traduction sont aussi des enzymes (et donc des ribozymes) mais ce n'est pas faux. Ils établissent des liaisons faibles avec différents substrats, catalysent la traduction, puis sont restitués intacts en fin de réaction.... ils ont donc bien les caractéristiques des enzymes. |
|
Quelques chiffres pour comprendre la complexité de ces enzymes
|
||||||||||||||||||||||
|
Les facteurs présentés ici sont ceux des ribosomes des procaryotes. Simplifié d'après EU - v10 - Un cycle de formation d'une liaison peptidique consomme 2 GTP et un ATP (pour lier l'aa à son ARNt). - Le premier aa de la chaîne (Met modifiée = fMet = N-formylméthionine) est enlevé par une enzyme qui raccourcit d'ailleurs souvent la chaîne de quelques aa. - Les ARNm sont lus dans le sens 5'->3'. - C'est un domaine particulier de la grande sous-unité ribosomiale (la peptidyl-transférase) qui catalyse l'établissement de la liaison peptidique. - On notera que le ribosome possède des sites de fixation de tous les cofacteurs, de l'ARNm et des complexes aa-ARNt.
40 % des antibiotiques connus inhibent le traduction (la tétracycline par exemple en se fixant sur un site de la petite sous-unité ribosomiale (30S) des procaryotes, empêche la fixation de l'aa-ARNt au premier site ribosomial). La traduction peut aussi être régulée à l'aide de facteurs empêchant l'initiation (chez les procaryotes par exemple les protéines ribosomiques inhibent la traduction de leur propres ARNm). |
|
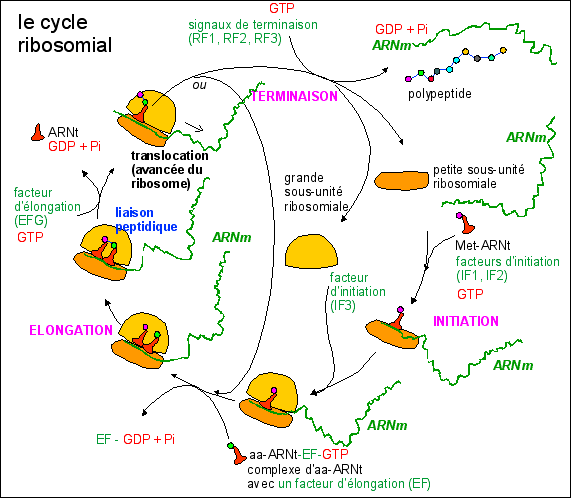
|
||||||||||||||||||||||||
|
Les ribosomes sont associés à un ARNm en polysomes. |
|
Un polysome se présente au MET comme une
chaîne (parfois fixés au
MET sous la forme d'une spirale) de ribosomes le
long d'une molécule d'ARNm qu'ils traduisent. |
|
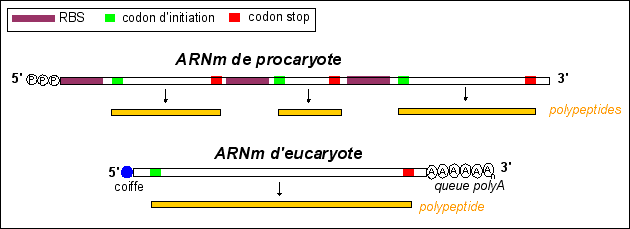
Les ARNm sont traduits plusieurs fois avant d'être détruits. Chez les procaryotes un ARNm contient souvent l'information pour plusieurs polypeptides différents alors que chez les eucaryotes chaque ARNm ne contient l'information génétique que d'un seul polypeptide (voir ci-dessous). Remarque: Les mitochondries et les chloroplastes possèdent des ribosomes qui réalisent la traduction des ARNm directement dans leur compartiment matriciel. De nombreuses protéines des ces compartiments possèdent des sous-unités codées à la fois par le génome nucléaire et le génome mitochondrial ou chloroplastique. |
|
La plupart des protéines sont organisées en complexes fonctionnels qui s'auto-assemblent sous le contrôle d'enzymes et de facteurs spécifiques (par exemple les molécules chaperons qui assurent le repliement de certaines protéines...).
Pour l'anecdote il existe une autre voie de synthèse de certains petits polypeptides qui ne passe pas par l'ADN et l'ARN mais cette voie semble très limitée (voir note).
|
||||||||||||||||||||
|
3.2.2 - La traduction suit un dictionnaire : le code génétique |
|
|
|
|
||||||||||||||||||||||
|
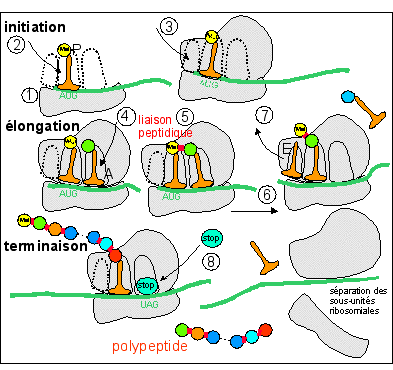
Chaque triplet de bases de l'ARNm (codon) est associé à un aa activé grâce à son ARNt spécifique. La correspondance entre les codons de l'ARNm et les aa constitue le code génétique. La traduction comporte trois phases : initiation, élongation et terminaison. Les ribosomes lient les aa correspondant à deux codons successifs puis progressent le long de l'ARNm en se décalant d'un codon.. La traduction est un mécanisme métabolique qui consomme de l'énergie (GTP) et qui est contrôlé par de nombreuses enzymes. La vitesse de traduction est lente : 2 à 4 aa par seconde pour les eucaryotes et jusqu'à 20 aa par seconde pour les procaryotes (à comparer aux 200 à 1000 nucléotides par seconde d'une réplication). Cependant, chez les procaryotes, transcription et traduction peuvent être simultanées (60 nucléotides/s = 20 codons/s = 20 aa/s), la traduction démarrant directement à l'extrêmité 5' de l'ARNm en cours de transcription (cf électronographies...). |
|
Le code génétique est universel, dégénéré et ponctué. Le code génétique est universel parce que tous les systèmes de traduction cellulaire utilisent les mêmes correspondances codon-aa. Cette universalité n'est pas absolue mais la rareté des exceptions (par exemple dans les mitochondries) prouve au contraire que ces exceptions sont plutôt des optimisations que des dérogations à la règle (il n'y a qu'une dizaine d'aa dans les peptides traduits dans la matrice mitochondriale et les ARNt mitochondriaux sont issus du génome mitochondrial) : tout changement a des conséquences très graves pour le sens de l'information génétique de la cellule. Le taux d'erreur de la traduction est de 10-4 à 10-3, soit pas plus d'un aa sur 1000 incorporé dans une protéine est incorrect. |
|
Le code génétique est dit dégénéré, car plusieurs codons correspondent à un aa (il existe 34 = 64 codons et 20 aa). Remarque: on dit aussi que le code est redondant mais ce terme est à éviter car cela voudrait dire que le code répète successivement deux informations identiques de façon différente....). Le code génétique est dit ponctué parcequ'il présente un codon initiateur (AUG ou GUG ayant une signification différente que lors de l'élongation), et trois codons stop (UGA, UAA, UAG). Remarque: en fait ce n'est pas le code qui est ponctué mais le message (l'information génétique contenue dans l'ARNm). Les codons ne sont jamais chevauchants et le message ne
contient aucun espace. |
|
Il est quasiment certain qu'il n'existe pas des ARNt spécifiques à chaque codon (possédant des anticodons complémentaires). Un type d'ARNt donné peut reconnaître plusieurs codons différents. Certains ARNt possèdent même au niveau de leur anti-codon une base adénine modifiée en inosine (hypoxanthine + ribose). Depuis 1966 où Francis Crick proposa le concept de l'appariement bancal (Wobble concept) les arguments s'accumulent en faveur de cette idée selon laquelle la base côté 5' est moins confinée et peut présenter un appariement bancal ( G-U ou G-C; C-G; A-U; U-A ou U-G; I-A ou I-U ou I-C). Chaque molécule d'ARNt peut alors reconnaître 4 codons différents. Il existe des ARNt modifiés qui sont capables d'insérer un aa en présence d'un codon stop (ils sont été très mal nommés ARNt supresseurs de mutation en supposant qu'ils sont capables d'empêcher qu'un codon STOP inséré de façon indue dans l'ARNm par mutation au niveau de l'ADN conduise à l'arrêt de la synthèse de la chaîne polypeptidique). |
||||||||||||||||||||
|
Compléments |
|
|
||||||||||||||||||||||||
|
|
|
Les étapes de la traduction sont les suivantes : INITIATION : (1) recrutement de l'ARNm
(extrêmité 5') par la petite sous-unité
ribosomiale et (2) fixation de l'ARNt initiateur
(portant une méthionine modifiée
(N-formylméthionine) : cet aa sera enlevé durant
ou après la synthèse polypeptidique) dans
le site P (codon initiateur = AUG ou
GUG) puis (3) fixation de la grosse
sous-unité; |
||||||||||||||||||||||||
| |
Chez les eucaryotes, sauf en de rares cas, chaque ARNm
n'est lu que d'une seule façon (le codon de
départ est AUG ou GUG et il n'y a pas de codon stop au
milieu de l'ARNm mais un seul en fin d'ARNm); on parle d'ARNm
monocistronique. |
|
Les ARNm des procaryotes possèdent un (ou plusieurs) site(s) de liaison du ribosome (ribosome-binding-site: RBS) situé (au minimum) à l'extrémité 5' (terminée par des groupes phosphate) dont la séquence est plus ou moins complémentaire de celle d'une portion d'ARNr de la grande sous-unité ribosomiale (16S). Plus la complémentarité des sites est grande, plus l'ARNm est lu. Les ARNm des eucaryotes possèdent une coiffe 5' (nucléotide guanine méthylé lié en 5'-5') reliée à l'extrêmité de l'ARNm par 3 groupes phosphate, qui est reconnue par le ribosome. Les ARNm eucaryotes ont aussi une queue polyA en 3' composée de nombreux nucléotides à adénine. |
|||||||||||||||||||||||
|
|
|
|||||||||||||||||||||||||
|
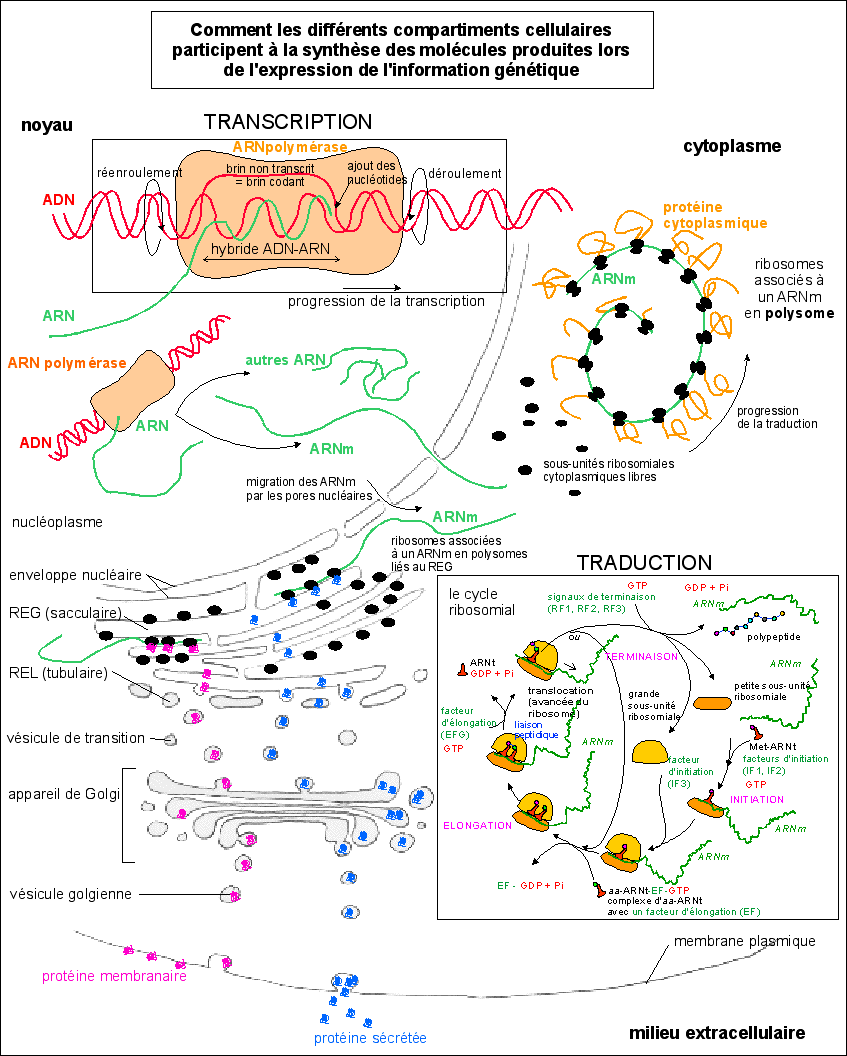
3.2.3 - Les peptides exportés subissent une maturation dans le REL puis dans l'appareil de Golgi avant d'atteindre la membrane ou d'être sécrétés par exocytose |
|
|
||||||||||||||||||||||||
|
||||||||||||||||||||||||||
|
Les peptides synthétisés dans le cytoplasme sont directement utilisés par la cellule pour le métabolisme (dans des enzymes...) ou pour des structures (histones...) (voir tableau). |
|
Les peptides synthétisés à la surface du REG migrent dans le REL (reticulum endoplasmique lisse, qui est tubulaire) puis dans l'appareil de Golgi et sont exportés dans des vésicules soit vers le membrane (protéines membranaires) soit vers l'extérieur de la cellule (protéines sécrétées par exocytose). |
|
C'est par exemple dans l'appareil de Golgi que sont accrochés les sucres (résidus glucidiques) des glycoprotéines (pour certains sucres, la glycosylation commence dans le REL). |
||||||||||||||||||||||
| |
|
Remarque : |
||||||||||||||||||||||||
|
4 - De l'information génétique au programme génétique |
|
|
||||||||||||||||||||||||
|
|
|
|
||||||||||||||||||||||||
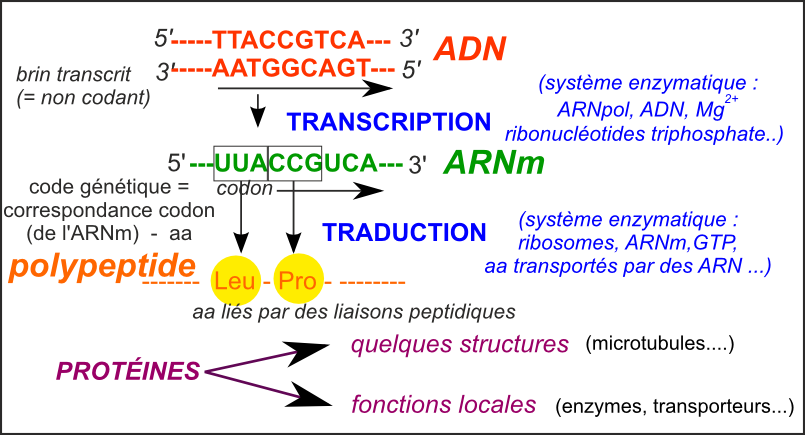 Ce bilan a comme principal avantage (scolaire) de faciliter la mémorisation du principe qui a guidé la compréhension des chercheurs dans les années 1960... Plus de 50 ans après, je crains qu'il ne soit plus qu'un leurre (du moins présenté ainsi). |
|
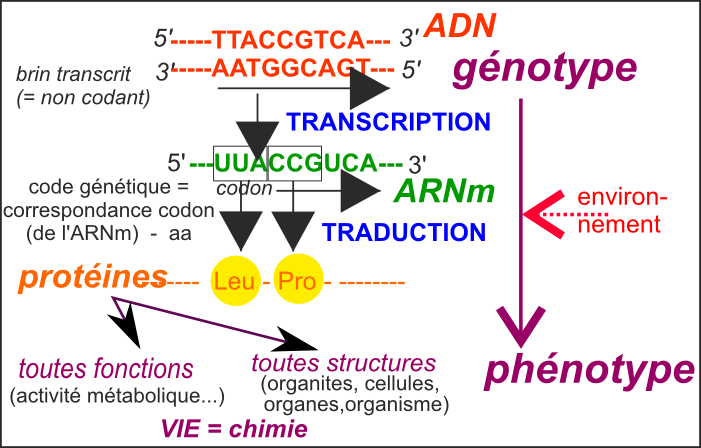 |
||||||||||||||||||||||||
|
L'information
génétique c'est d'abord la séquence
des nucléotides de l'ADN. C'est une information
linéaire. Entres les années 1960 et les années 1990
on s'est appuyé sur cette analogie entre un texte
écrit (message) et la fonction métabolique des
protéines (l'ADN enfermait le
message originel, l'ARN était une transcription
lisible, et les protéines constituaient le message
utilisable par la cellule). L'ensemble des gènes moléculaires constitue le génome. Un gène moléculaire est une unité de fonction : une séquence d'ADN codant pour un produit (ARN et/ou peptide). Les notions de génotype et phénotype sont chromosomiques et héréditaires. Le génotype étant le liste des allèles d'un organisme. Les allèles étant les diverses formes des gènes héréditaires. Les gènes héréditaires étant les portions de chromosomes qui gouvernent l'apparition d'un caractère héréditaire. Le phénotype résume l'ensemble des caractères héréditaires visibles (et non tous les caractères visibles). |
|
La théorie a rigidifié la liaison ADN-protéines en tentant de fusionner les notions de génotype et phénotype, issues de l'hérédité avec la biologie moléculaire (voir plus bas) : L'information génétique est contenue dans l'ADN (figée et stable), copiée exactement (sans erreur ni modification) dans l'ARNm, puis lue grâce aux ribosomes pour être exprimée dans des polypeptides en respectant fidèlement le code génétique universel. L'ARN n'est qu'un intermédiaire et les polypeptides sont le seul message de l'ADN. Les protéines déterminent le phénotype (moléculaire mais aussi cellulaire, puis au niveau de l'organisme). Les protéines sont le seul moyen d'expression du génotype. Les protéines expliquent tout phénotype. L'environnement désigne un facteur flou externe au système d'information qui explique tout ce qui ne colle pas avec la liaison rigide génotype-phénotype. En tentant de fusionner les notions de gène
moléculaire et de gène héréditaire
la théorie de l'information génétique
mélange les vocabulaires hérédiatires et
moléculaires: |
||||||||||||||||||||||||
|
Vers un changement profond de compréhension 5 - L'organisation du génome est complexe et mal connue |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Un gène moléculaire est défini par sa nature chimique (ADN), sa séquence (suite de monomères) et sa fonction (servir de copie lors de la transcription). |
|
5.1- Les gènes sont des unités de fonction |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
Si l'on considère que les gènes moléculaires contiennent une information, cette information est linéaire. De plus, comme la seule fonction connue précisément de l'ADN, découpé en gènes moléculaires, est une fonction passive: servir de modèle lors de la transcription, l'information génétique est donc une information pour une molécule (d'ARN). |
|
.Les gènes
moléculaires sont donc
des segments
d'ADN contenant une information
linéaire copiée
dans une molécule de type ARN lors de la
transcription
puis, pour les ARNm, cette information est traduite
ou exprimée
dans une molécule peptidique. Le contrôle de l'expression de l'information génétique peut se faire au niveau de la transcription, de la maturation des ARNm ou de la traduction. |
|
Les gènes sont des unités fonctionnelles de l'information génétique.
L'information
génétique est la séquence
de l'ADN ou de l'ARN ou encore des
protéines. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
pages sur les génomes (en cours de rédaction) |
|
Le nombre de gènes moléculaires des différentes cellules vivantes n'est pas précisément connu. Un gène moléculaire peut correspondre à plusieurs produits et inversement un produit peut nécessiter plusieurs gènes moléculaires, les gènes moléculaires peuvent se chevaucher... bref, ceci est une autre histoire: celle de la génomique (science des gènomes) ou biologie moléculaire du gène moléculaire. |
|
Une nouvelle branche de la génomique se développe : la protéomique qui s'intéresse aux seuls gènes moléculaires contenant une information pour des protéines. On utilise aussi le terme de transcriptome pour désigner l'ensemble des gènes transcrits et de protéome pour désigner ceux qui sont transcrits puis traduits. |
|
Malgré le séquençage complet du génome humain on n'espère au mieux que 25.000 gènes moléculaires associés à des protéines. Si on ajoute les quelques 2.000 gènes moléculaires associés à des ARN non traduits cela fait un peu court pour que l'imaginaire continue de voir dans les protéines autre chose qu'un support matériel de la vie. Quand au "pouvoir magique" du gène moléculaire il s'estompe petit à petit. |
||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
On remarquera qu'il existe une idée fausse qui est souvent propagée qui consiste à dire que plus un organisme est complexe plus il a de gènes. Au contraire le nombre de gènes ESTIMÉ semble assez fixe pour un type donné de cellule. |
|
|
|
Les unicellulaires (procaryotes ou eucaryotes) ont donc un nombre de gènes approximativement compris entre 3.000 et 6.000) alors que les pluricellulaires (forcément eucaryotes) ont entre 12.000 et 25.000 gènes. |
||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
5.2 - Plus les organismes sont complexes plus leur ADN comporte des gènes morcelés (par des introns), une grande quantité d'ADN non codant, et plus la liaison gène-protéine est complexe |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Le génome des procaryotes contient une très petite quantité d'ADN non codant alors que la majorité du génome des eucaryotes est non codant.
|
|
Un gène au sens strict doit être
associé à un produit (ARN fonctionnel : ARNm,
ARNr, ARNt....). On dit que l'ADN est alors codant.
Il existe une grande quantité de l'ADN qui n'est pas
codant, ne serait-ce que l'ADN des introns (mais
on a vu que les limites d'un intron peuvent être
fluctuantes et que l'ADN d'un intron peut alors être
codant). Il y a une grande difficulté à étudier le génome étant donné sa taille et sa complexité: les chiffres donnés ici ne sont pas définitifs.... j'ai indiqué quelques résultats du programme ENCODE...
Pour des détails voir page sur les génomes |
|
TOTAUX
estimés |
||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Un nouveau mode
de communication : |
|
Dans un article historique dans Nature de 2006 traduit
ici: Qu'est-ce
qu'un gène ? Helen
Pearson soulignait déjà les résultats
publiés en 2005 (réf 7
et 8) selon lesquels 63% du génome
(au sens de la totalité de l'ADN)
de la souris serait effectivement TRANSCRIT, de
façon continue, en donnant des ARN de taille
très variable. Cette énorme masse d'ARN
"non-sens" posait d'intéressantes questions. Certains
considèraient déjà que le gène est
désormais défini par une séquence d'ARN
et non d'ADN.
LR 471, janvier 2013, pp46-49 ENCODE Project Writes Eulogy For Junk
DNA , E. Pennisi, Science, 337, 1159, sep 2012 (article
en anglais au 5/01 |
|
Les premiers résultats publiés dans Nature en septembre 2012 confirment l'idée selon laquelle une grande partie du génome humain est actif malgré l'absence de gènes en son sein. L'activité de l'ADN est considérée
selon plusieurs niveaux : Avec ces critères, 80% de l'ADN des cellules humaines étudiées est actif biochimiquement - la traque a porté sur 147 types cellulaires humains (cellules du sang, du foie, des neurones, des os, embryonnaires...). |
|
Quelques résultats: Je précise qu'étant donné la technicité des résultats il est quasiment impossible à un non spécialiste d'en tirer des éléments synthétiques. Il faudra attendre une vraie vulgarisation. |
||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
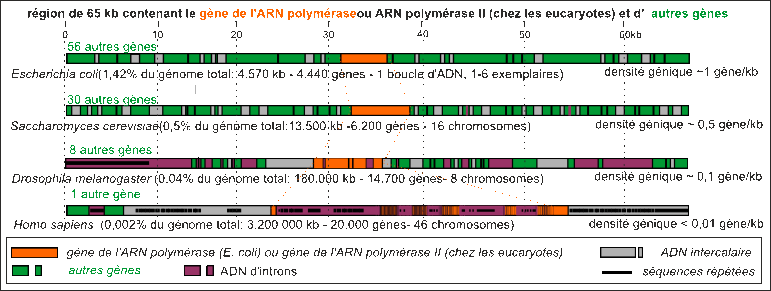
Les gènes des procaryotes ne contiennent pas de séquences non codantes (qui sont alors placées entre les gènes) alors que les gènes des eucaryotes contiennent un nombre de séquences non codantes d'autant plus grand que l'organisme contient de nombreux types cellulaires. |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
La densité génique est le
nombre de gènes / taille du génome (cette
représentation ne tient pas compte des gènes
chevauchants puisque vous savez que les 2 brins de l'ADN
peuvent être codants; dans la région de l'ADN
étudiée on considère qu'il n'y a pas de
gènes portés par les deux brins
séparément). |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |
|
La première hypothèse, la plus évidente, serait alors que le morcellement augmente lors de chaque division. Plus un organisme possède de cellules plus son ADN serait fragmenté. Cela reste à démontrer. Corrélativement il ne semble pas (même si l'exploration précise n'a sans doute jamais été faite) qu'entre une cellule embryonnaire et une cellule différenciée la masse d'ADN soit si différente, du moins de façon constante et avérée (on rapporte de nombreux cas de polyploïdies ou au contraire d'ADN manquant, dans les cellules différenciées, mais aucune généralisation ne peut être faite actuellement). |
|
Une autre idée séduisante est celle d'un résultat de l'évolution : les organismes les plus simples étant supposés être apparus d'abord, la complexité des génomes reflétant alors l'évolution des organismes. Mais, attention, il ne s'agit pas ici d'évolution par ajout ou modification de gènes, mais bien par ajout des séquences intercalaires et/ou répétées... Il est clair que ce ne sont pas les organismes les plus évolués (au sens de distance phylogénétique maximale et non d'éloignement dans le temps) qui ont le plus de gènes, mais de ceux dont le génome est le plus complexe. |
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Un essai de mise-à-jour indispensable 6 - L'expression stochastique (ou aléatoire) des gènes (ESG) |
ancienne page (1ES-1L) liaison génotype-phénotype |
|||||||||||||||||||||||||||||||
|
« Ce n'est pas tant la précision des prédictions génétiques qui fait problème que leur caractère empirique, leur bas niveau théorique. Il n'y a en effet pas de théorie biologique générale qui permette de passer du génotype au phénotype.» Jean Gayon, 2011, Le Hasard au cœur de la cellule, Ed. Matériologiques, ch 4 p 290 |
|
Les théories qui s'affrontent quant à la signification de l'information génétique sont davantage des théories du vivant, qui englobent donc l'évolution, qui est au centre de la biologie depuis plus d'un siécle, plutôt que des théories génétiques. |
|
J'ai fait le choix de présenter d'abord les résultats incontournables de la biologie moléculaire avant de souligner les problèmes liés à la présentation des manuels scolaires qui se sont TOUS cantonnés à la théorie de l'information génétique. Quelques ouvertures complètent le chapitre. |
||||||||||||||||||||||||||||
|
|
|
Cette compréhension est récente et remet en cause une vision figée et statique du génome (la version détermiste de l'ordre par l'ordre, celle du programme génétique - voir plus bas), mais elle repose sur d'innombrables faits expérimentaux et il est plus que temps qu'elle soit enseignée. Ce n'est pas une théorie mais bien un ensemble de données, dont certaines très anciennes, formant une idée incontournable,mais nouvelle, dans son acceptation par la communauté scientifique : l'aléatoire* se trouve au centre de l'expression de l'information génétique. Ce qui devrait conduire à revoir la notion même d'information génétique. Cependant, cette vision, pour très scientifique qu'elle soit, n'en est pas moins diffusée EN MÊME TEMPS que le darwinisme cellulaire de Kupiec qui a un toute autre dimension, philosophique notamment. Je lui consacre un autre paragraphe. |
||||||||||||||||||||||||||||||
|
Le problème théorique premier est celui de la liaison gène-protéine dont la complexité ne cesse d'augmenter. On a pris l'habitude de parler de "l'expression de l'information génétique" en supposant que l'information est dans l'ADN; ce qui est de plus en plus discutable. Le gène pourrait être défini pa l'ARN, l'information génétique se trouvant alors aussi bien au niveau de l'ADN que de l'ARN ou des protéines. La complexité augmente, même pour les
procaryotes. La complexité de l'expression de l'information génétique ne sera pas résolue en multipliant à l'infini les molécules contrôlant tel ou tel processus au sein d'un réseau de plus en plus complexe. Il est nécessaire de changer radicalement les fondements mêmes de la biologie moléculaire programmiste ou déterministe. |
|
Comme il n'est pas question ici de présenter ne serait-ce qu'un résumé de cette complexité je voudrais juste montrer une voie dans laquelle de nombreux laboratoires de biologie moléculaire français se sont engagés, notamment autour de Jean-Jacques Kupiec : la voie (ou la théorie) de l'expression aléatoire des gènes ou expression stochastique* des gènes (ESG), théorie qui se propose de remplacer la théorie du programme génétique (dont nous parlerons ensuite). |
|
Sources: |
||||||||||||||||||||||||||||
| |
|
|
||||||||||||||||||||||||||||||
|
*aléatoire, stochastique, hasard : une remarque épistémologique stochastique = qui contient une variable aléatoire = qui est au moins partiellement du au hasard |
|
Dans sa définition courante le hasard
n'est qu'une absence de cause connue, c'est paradoxalement un
principe explicatif par un aveu d'ignorance. Certains lui
confèrent les attributs de la finalité dans une
confusion relativiste (en affirmant l'absence de cause finale,
ils lui susbtituent le hasard !!!). (Les
épistémologues modernes se démarquent
d'une conception classique du hasard qu'ils
préfèrent nommer hasard absolu, lié
à l'absence de cause (ce qui est absurde pour un
réaliste); les notions complexes (et ayant
évolué au cours de l'histoire) de
déterminisme, contingence, immanence, ordre.... sont
souvent revisitées de façon pas toujours
très claire et on a bien du mal à comprendre les
positions épistémologiques des uns et des
autres). En m'attachant aux causes, et à des
définitions claires de ces causes, je pense que l'on
peut arriver à comprendre les positions des autres sans
soi-même sombrer dans la confusion. La
stochasticité revendiquée est celle d'une
variation inhérente au vivant qui n'empêche en
rien une causalité matérielle et formelle. |
La réflexion de la jeune épistémologue Francesca Merlin autour des notions de hasard utilisées par les biologistes modernes est une première tentative de clarifier le débat : ce qu'elle nomme "le hasard au sens faible" est l'utilisation moléculaire de l'interprétation probabiliste, c'est tout à fait probant; par contre, ce qu'elle nomme "le hasard évolutionnaire", qui serait la version utilisée dans la théorie darwinienne moderne (Théorie Synthétique Moderne) pour désigner la relation variation-sélection-adaptation, laisse bien davantage de questions étant donné qu'elle commence par séparer ce qu'elle appelle un hasard objectif et un hasard subjectif. Son discours (sa thèse est disponible ici) est aussi peu clair, à mon avis, sur la fin et les causes : on ne peut pas faire l'impasse sur une discussion profonde des thèses évolutives qui s'affrontent (on est loin d'avoir l'unanimité) et pour cela il faut d'abord les lister, travail qui n'a pas été fait. (Elle rédige le chapitre 7 de HCC, Francesca Merlin : Pour une interprétation objective des probabilités dans les modèles stochastiques de l'expression génétique, pp215-252, voir ci-dessus) |
|||||||||||||||||||||||||||||
|
Article fondateur : Stochastic Gene Expression in a Single Cell, Michael B. Elowitz et al., Science, 297,1183 (2002)
« L'expression aléatoire des gènes est un phénomène répandu dans le monde vivant, modélisable en plusieurs composantes, explicable par des indices moléculaires et topologiques, parfois transmissible et héritable, manifestement contrôlé.» Heams, HCC, p 47 |
|
La transcription commence au niveau d'un promoteur. Certaines de ces séquences, isolées à partir de phages (virus de type bactériophages), peuvent forcer la transcription de gènes étrangers dans le génome d'un hôte. On sait donc insérer des gènes d'eucaryotes avec un promoteur viral très efficace dans certaines cellules. |
|
« L'organisme sur laquelle l'équipe d'Elowitz travaille est une bactérie bien connue des biologistes : Escherichia coli. Pour mettre en évidence une éventuelle stochasticité dans l'expression, ils intègrent, dans le génome de la bactérie, deux gènes « rapporteurs », c'est-à-dire codant chacun pour deux protéines de fluorescences différentes. Ils les placent dans des conditions permettant d'espérer un niveau d'expression identique (positions symétriques par rapport à l'origine de réplication, promoteurs identiques). Leur prédiction est alors la suivante. Soit chaque bactérie exprime effectivement ces gènes dans des proportions identiques, et dans ce cas la fluorescence résultante sera la même dans toutes les bactéries : toutes les bactéries auront peu ou prou la même « couleur ». Soit chaque bactérie exprime ces gènes de manière aléatoire, de sorte que chacune exprime un ratio des protéines correspondantes qui lui est propre ; dans ce cas, la « couleur » résultante variera de l'une à l'autre. Cela serait alors le signe que, quand bien même ces bactéries possèdent exactement le même génome, et quand bien même elles sont dans un environnement commun, elles répondent différemment à cet environnement, de manière stochastique. Comme le titre de l'article le laissait percevoir, c'est le second cas de figure qu'il observe par défaut. L'article fera la une de Science.» Heams, HCC, p 38 |
||||||||||||||||||||||||||||
| |
La démonstration d'Elowitz repose sur un
grand nombre de présupposés
théorico-pratiques qu'il ne serait pas inutile de
préciser. Si le présupposé du
déterminisme génétique classique tombe
avec cet article il n'en reste pas moins que de nombreux
autres restent forts. |
|||||||||||||||||||||||||||||||
|
Ceux qui travaillent dans le nouveau paradigme stochastique tendent à généraliser cette vision et poussent leur raisonnement plus loin : d'après Paldi, 2007 (in Génétiquement indéterminé)
Pour des données de
recherche voir par exemple la page
STOCHAGÈNE |
|
Dans le modèle déterministe - qui voulait qu'un gène contienne une information libérée lors de la transcription - on n'a cessé de trouver de nouvelles séquences d'ADN et des protéines régulatrices associées à l'expression de la séquence transcrite apellée gène (séquences promoteur, "enhanceurs", ""insulateurs"... sans compter, par exemple, les 70 protéines indispensables au fonctionnement du complexe ARN-polymérase...). La complexité de l'expression de l'information génétique est devenue telle qu'il est devenu impossible de prévoir dans un environnement donné si un gène sera transcrit ou non. Cette complexité est aussi associée, il ne faut pas l'oublier, à la nécessité de prendre en compte le faible nombre de molécules impliquées dans ces processus (deux copies du gène au plus, quelques molécules à quelques centaines de molécules pour les systèmes enzymatiques...«80 % des protéines présentes dans une bactérie le sont à moins de 100 exemplaires » (HCCp42)) ce qui exclue les interprétations chimiques classiques basées sur les lois des espèces en solution (loi d'action de masse...). Et le nucléoplasme est tout sauf une solution. On doit donc y ajouter la structuration du nucléoplasme (voir cytologie dans le chapitre précédent) et la nécessité de canaliser les réactions pour atteindre les vitesses constatées. Le deuxième problème consiste dans l'accessibilité
de l'ADN chez les eucaryotes. Quelle que soit sa
forme - au sein de l'hétéro- ou de
l'euchromatine - dans le noyau, l'ADN est toujours sous la
forme d'un nucléofilament enroulé
périodiquement autour des nucléosomes (structure
en "collier de perles"). Comment imaginer q'une
séquence régulatrice spécifique puisse
être accessible à des protéines autrement
qu'en imaginant qu'il existe des systèmes qui rendent
accessible cette même séquence ? Le raisonnement
circulaire peut être brisé par la
considération de l'agitation moléculaire
permanente qui fait que les structures sont des assemblages
dynamiques en perpétuel remaniement (la structure de
l'hétérochromatine est considérée
comme ayant une demie-vie de quelques minutes, alors que
l'assemblage d'un facteur de transcription au sein d'un
complexe ne reste stable que quelques secondes...). L'accessibilité
d'une séquence est donc plus ou moins grande en
fonction de la stabilité des assemblages qui la
rendent ou non accessible, mais elle n'est jamais nulle.
Cette considération a donc rapidement conduit les
biologistes moléculaires sur la voie de l'analyse
probabiliste des phénomènes. Tout
gène est potentiellement transcrit mais avec une
probabilité variable. (Les gènes transcrits en
très faible quantité dans une cellule sont
l'objet de ce que l'on apelle la "transcription
illégitime"). Le contrôle de la transcription
dans une lignée cellulaire est alors
transféré pour une bonne part sur les
modifications de l'ADN qualifiées d'épigénétiques
(acétylation, phosphorylation, poly-ADP-ribosylation et
méthylation...) qui permettraient de marquer un
gène qui vient d'être transcrit ou les histones
voisines de ce gène en fonction du métabolisme
de la cellule. On
ne peut s'empêcher de penser que cette pirouette
probabiliste qui permet de jusifier un déterminisme
expérimental (du fait que la corrélation
existe toujours à l'échelle cellulaire entre
la présence d'un facteur de transcription et la
transcription d'un gène) repose en fait sur un
indéterminisme expérimental
(déterminé mais non prévsible) et donc
en dernier lieu sur une variation. Il ne manque pas de voix pour affirmer que, finalement, cette stochasticité ne change rien au déterminisme génétique, puisqu'à l'échelle cellulaire le rôle des gènes est préservé. Cette position qui notamment assimile variation et bruit (et refuse de considérer l'individualité de chaque cellule) ne tient pas à l'analyse (voir Amzallag). C'est aussi pour cette raison que je vais développer dans le chapitre suivant les incohérences de la position classique toujours enseignée. |
||||||||||||||||||||||||||||||
|
7 - Critique des rôles de l'ADN imaginés dans la théorie de l'information génétique |
|
|||||||||||||||||||||||||||||||
|
|
|
Les faiblesses de la
vision de la théorie de l'information ou du programme
génétique : |
|
|
||||||||||||||||||||||||||||
| |
|
7.1 - Un premier exemple d'excès de la théorie : la notion de programme génétique |
||||||||||||||||||||||||||||||
 extrait du film "Le cinquième élément" «200 milliards de mémogroupes d'ADN, bien suffisant pour faire un être vivant; l'homme en contient 40...» L'incroyable Hulk « l'ADN : la structure fondamentale de la vie...» Spiderman 1 « quelques bases modifiées = nouvelle espèce....» Underworld 1 « L'immortalité conférée par une bactérie-virus...» |
|
On est sans conteste resté dans les médias et dans de très nombreuses présentations scolaires à ces raisonnements de science-fiction. Quelques affirmations dans cette veine : - L'ADN contient toute l'information pour faire un être vivant ; c'est laisser de côté toute la complexité de la génèse, la transmission et l'expression de cette information plus ou moins modifiable et supposer, dans la forme la plus poussée de l'énoncé, que la vie soit réduite à des interactions matérielles (réductionnisme moléculaire); affirmation corrigée : l'ADN contient l'information nécessaire pour faire les protéines du vivant. |
|
- Chaque cellule d'un pluricellulaire possède la totalité de l'information génétique de l'individu mais n'exprime qu'une petite partie de cette information. Toutes les cellules d'un individu pluricellulaire ont la même information génétique : le programme génétique. Pour justifier le fait que la vie réside dans chacune des cellules du pluricellulaire on est bien obligé d'imaginer que le programme génétique (qui gouvernerait la vie) est complet dans chaque cellule; ceci est loin de cadrer avec les résultats actuels. L'information génétique n'est qu'une information pour des molécules affirmation corrigée : Chaque cellule d'un pluricellulaire hérite lors de la division cellulaire de l'ADN de la cellule mère qui a été repliqué avant la division. Cet ADN peut être incomplet et être modifié ensuite par chaque cellule. |
|
- L'ADN contient l'information spécifique de chaque individu, ce qui nous fait différer de tous les autres (la diversité des individus c'est la diversité de leurs gènes) ; c'est accorder bien trop d'importance au niveau moléculaire; la vie n'est pas définie en dessous du niveau cellulaire; loin de nous différencier, la (bio)chimie nous unit ; affirmation corrigée : les gènes sont presque identiques au sein d'une espèce mais certaines séquences répétées d'ADN non codant permettent de différencier les individus de la même espèce. On trouve partout dans les médias un concept de gène qui est totalement erroné : les gènes d'un comportement, les gènes d'une capacité intellectuelle, voire les gènes d'une maladie (la maladie génétique, dont l'origine se trouve uniquement dans une modification génétique est extrêmement rare). |
||||||||||||||||||||||||||
| |
La théorie de l'information génétique postule donc à la fois que la vie est strictement matérielle et que l'ADN contient toute l'information nécessaire à la vie. On est passé clairement ici à une position philosophique (matérialiste) de la conception de la vie. |
|
Le patrimoine génétique est une expression qui doit désigner uniquement l'information génétique contenue dans l'ADN pour faire des protéines, en tant qu'elle est transmise lors de la reproduction. C'est la part héritée de l'ADN d'une cellule. Au niveau d'un organisme la notion est nettement plus floue et on devrait éviter de l'employer. |
|
L'information génétique est cellulaire La notion d'information génétique, information pour des molécules, doit se limiter au niveau d'une cellule. L'information génétique d'un organisme pluricellulaire est un concept flou à ne pas employer. |
|||||||||||||||||||||||||||
| |
|
Remarque: |
|
Certains se défendent d'être matérialistes au sens philosophique du terme et prétendent qu'il existerait un matérialisme "scientifique", voire "expérimental": une position selon laquelle le scientifique, qui n'aurait accès qu'à la matière par l'expérience, devrait se contenter d'une interprétation matérielle. Cette position est TROMPEUSE. Soit la vie est un phénomène qui dépasse la matière et c'est le sujet de la biologie, soit la vie est strictement matérielle et dans ce cas il n'y a plus de biologie mais une chimie du vivant qui n'est qu'une partie de LA chimie. Voir Qu'est-ce que la vie ? et surtout la page d'André Pichot sur l'Histoire de la notion de vie. |
||||||||||||||||||||||||||||
| |
|
7.2 - Un deuxième exemple d'excès de la théorie : les mutations seules sources de variation de l'information génétique |
||||||||||||||||||||||||||||||
|
Extrait du génériquede X-men 1, ou du générique de fin de X-Men 2 (identiques) « la mutation, la clef de notre évolution....» |
|
Le premier problème vient de ce que l'on a figé l'information génétique dans la théorie. Au lieu d'en faire une information dynamique comme nous le suggère toujours le vivant on l'a figé en un CODE. C'est une autre appellation du programme supposé être contenu dans l'ADN. |
|
Il suffit de voir combien de personnes, et même des chercheurs, voire des pédagogues, utilisent le mot "code génétique" à la place d'information génétique. ( Par exemple la conférence de Jean-Jacques Kupiec à l'ENS, chercheur que l'on peut difficilement taxer de légéreté... et qui s'efforce justement de lutter contre l'idée de programme génétique). |
|
On trouve dans BMG (p414) qui est un livre de génétique de fort bon niveau (1er et 2ème cycles universitaires) le titre "Trois types de mutations ponctuelles altèrent le code génétique". Il est clair que ce n'est en aucun cas le code génétique qui est altéré - mais c'est parce que le code est au contraire inchangé qu'il y a répercussion éventuelle d'un changement au niveau d'un polypeptide lorsqu'il y a un changement dans l'ADN. |
||||||||||||||||||||||||||
|
page simplifiée sur les mutations (ancien cours de seconde) Page compémentaire d'ouvertures sur les mutations et la variation |
|
Dans la théorie du programme génétique la seule source de variation de l'information génétique sont les mutations. Car, étant donné que le programme réside dans l'ADN, la seule source de variation doit se trouver dans l'ADN : ce sont les mutations. Comme on a rapidement
trouvé des modifications très complexes de l'ADN
avec |
|
Voici un petit aperçu de ce que l'on trouve encore dans tous les livres.
Une mutation ponctuelle est
tout simplement le changement d'une seule base (mais
le raisonnement peut être étendu
à quelques bases) par modification,
délétion ou insertion. On distingue donc trois types de "mutations
ponctuelles" selon leurs conséquences: Les mutations ne se font pas au hasard ! Les agents mutagènes (u.v., substances chimiques) agissent au contraire sur des bases spécifiques et accessibles dans des zones non réparées.... bref, tout le contraire d'un mécanisme aléatoire. On lit même parfois que les modifications génétiques faites par génie génétique (insertion d'un gène...) sont des mutations.... c'est encore un abus, la mutation doit être naturelle, spontanée ou induite. (Sinon il est évident que pour cette manipulation génétque on est sûr du mécanisme, et qu'on renforce alors de façon indue l'idée que les vraies mutations sont des phénomènes de modfication de l'ADN !). |
||||||||||||||||||||||||||||
| |
|
Le second problème vient de la méthode que l'on a utilisée pour étudier les mutations : la sélection. On laisse agir des agents mutagènes (u.v., substances chimiques...) sur une population et on sélectionne ensuite les individus qui résistent (par exemple à un antibiotique). Ces individus résistants sont qualifiés de mutants. Il est évident que leur résistance peut reposer sur de très nombreux mécanismes (voir mutations). On mesure non pas une résistance à un facteur environnemental mais la survie de quelques individus plus ou moins préadaptés. |
|
Remarque: |
||||||||||||||||||||||||||||
| |
|
7.3 - Un troisième exemple d'excès de la théorie : la (con)fusion entre gène héréditaire et gène moléculaire |
||||||||||||||||||||||||||||||
|
Un peu d'histoire...
Quelques pages d'histoire: |
|
Le terme de gène désigne
à partir de 1910 les particules
héréditaires postulées par les
hybrideurs au XIXème. |
|
On peut dater de 1909 l'apellation des particules héréditaires comme "gènes" par le biologiste danois Wilhem Johannsen (1857-1927) (qui proposera aussi phénotype et génotype). Mais la notion de particule héréditaire transmise lors de la reproduction et qui porterait les "traits de caractères" héréditaires est bien antérieure. En 1866, le moine tchèque, Johann Mendel (1822-1884) publie ses résultats sur des expériences menées chez le pois (Pisum sativum). Hugo de Vries (1848-1935) qui travaille sur Oenothera lamarckiana (et nomme mutations les changements brusques de caractères observés dans la descendance qui seraient à l'origine de nouvelles espèces) les nomme pangènes. Le terme d'allèle semble dater de 1902 , utilisé par William Bateson qà qui l'on doitr aussi les termes d'homozygote, d'hétérozygote ou de génétique (1905); dans ce tout début du XXème la génétique devient une science à la mode et des chaires universitaires de génétique sont créées). Il semble que ce soit Sutton (1903) et Boveri (1904) qui proposèrent pour la première fois d'associer les gènes aux chromosomes qui deviendraient ainsi supports de l'hérédité. |
|
Thomas Morgan travaille sur les mutants de la mouche du vinaigre : Drosophila melanogaster. Après s'être opposé à la théorie chromosomique de l'hérédité, il en deviendra le fervent défenseur. Morgan obtient un mutant mâle aux yeux blancs dans une population aux yeux rouges (caractère sauvage). Comme ce trait de caractère n'apparaît que chez le mâle, l'idée lui vient de l'associer à un chromosome sexuel car il existe, chez la drosophile comme chez l'homme, un déterminisme chromosomique du sexe: les femelles possédant une paire (XX) d'homologues (confirmé par N. Stevens, élève de Morgan, en 1909) et les mâles un chromosome X et un chromosome Y. Morgan découvre avec ses collaborateurs d'autres mutations dont ils étudient la transmission héréditaire. Elles forment 4 groupes de liaison: c'est-à-dire qu'elles ne se répartissent pas au hasard dans les descendants mais qu'elles présentent une liaison (linkage, en anglais): elles ont tendance à rester associées. Ils émettent alors l'hypothèse que ces quatre groupes de liaison sont assimilables aux 4 paires de chromosomes de la drosophile. La liaison étant donc "simplement le résultat mécanique de la localisation des gènes dans les chromosomes" . Les gènes deviennent alors des unités de mutation et de recombinaison: un gène est une unité mutable appartenant à un groupe de liaison (statistique) : le chromosome. Les gènes sont cartographiés grâce à l'étude des mutations et l'on propose une disposition linéaire des gènes le long du chromosome. |
||||||||||||||||||||||||||
| |
|
Un peu avant la seconde moitié du XXème siècle des physiciens et des chimistes venus à la biologie et en étudiant principalement des virus et des bactéries, vont développer la biologie moléculaire du gène et associer l'ADN à des enzymes reliées elles-mêmes à différentes propriétés chimiques des bactéries. L'ADN devient la molécule qui transporte une information génétique et les protéines vont devenir les éléments majeurs du phénotype. La théorie de l'information génétique se met en place. |
|
Quelques pages d'histoire: G. Beadle, E. Tatum et le concept un gène - une enzyme À propos des travaux de F. Griffith (1928) et d'Avery, McLeod, McCarthy (1944) sur les pneumocoques |
||||||||||||||||||||||||||||
|
Pour une nouvelle compréhension de l'hérédité, en liaison avec l'évolution |
|
Près de un demi-siècle plus tard ces deux
visions (celle du gène héréditaire,
particule héréditaire - et celle du gène
moléculaire, information pour une molécule) ne
sont toujours pas unifiées. Il y donc vraiment un
fossé entre ces deux visions. Le principal
obstacle vient de la différence d'ordre de grandeur
entre le gène moléculaire (quelques 1-10 kb) et
le fragment de chromosomes (quelques 1000-10.000 kb).
Ce qui
est sûr Il faut éviter de mélanger le vocabulaire de la génétique héréditaire (génotype, phénotype, allèle, caractère) avec celui de la génétique moléculaire (séquence d'un gène, information génétique, isoformes...). Mais le mal est fait et les confusions se trouvent partout. À chacun de rester vigilants en précisant - lorsqu'il emploie les mots génétique ou héréditaire ou gène... - de quoi il parle !!! |
Un bon article (court et simple) utilisable en classe qui présente correctement le lien entre trois gènes associés et la maladie d'Alzheimer. (version formatée, version texte html): Marie-Laure Théodule " Maladie d'Alzheimer : trois nouveaux gènes identifiés " La Recherche n° 435, novembre 2009, pp 8-10 |
|||||||||||||||||||||||||||||
|
Conclusion: le mal est fait : le vocabulaire de la théorie chromosomique de l'hérédité à été fusionné indument avec celui de la biologie moléculaire; la seule solution est de rester attentif à ce dont on parle. |
|
|
||||||||||||||||||||||||||||||
|
8 - Vers de nouvelles théories du vivant |
|
En travaux |
||||||||||||||||||||||||||||||
| |
|
8.1 - L'ontophylogénèse : une théorie darwinienne de la lignée cellulaire |
||||||||||||||||||||||||||||||
|
J'ai tenté dans la page sur l'évolution de présenter en quoi cette théorie était davantage une théorie évolutive du vivant qu'une simple théorie génétique. Ici, je veux simplement montrer en quoi cette théorie modifie la perception que l'on a de l'information génétique. |
|
Dès 1983 J-J. Kupiec avait proposé un mécanisme aléatoire de différenciation cellulaire. Depuis, il n'a cessé de le peaufiner en l'intégrant dans une perspective "darwinienne" de sélection. Mais il est clair pour moi que cette perspective nous fait sortir du domaine de l'expérimental. |
|
« Le processus de variation-sélection se fait en deux phases : la première, très rapide, est indétectable et consiste dans une variation suite à un stimulus, la seconde, l'adaptation à l'environnement, est plus lente.» (Heams) [on voit ici encore combien le darwinisme est une théorie de l'adaptation et non de la variation, qui repose bien trop souvent sur la mutation] |
|
Kupiec (1983) A probabilist theory for cell
differentiation, embryonic mortality and DNA C-value paradox,
Speculations in Science and Technology, Vol. 6, No 5 |
||||||||||||||||||||||||||
|
Pour montrer comment il est facile de dériver en invoquant une "sélection naturelle" pour décrire un équilibre bistable voici un texte de Heams. La sélection naturelle ressort de l'invocation consensuelle, politiquement correcte, mais n'est en rien nécessaire à la compréhension du phénomène. Le hasard non plus.
De même, rien à redire sur la conclusion de
Heams jusqu'aux mots solution et mémoire
qui sont franchement inadaptés après avoir
rejeté la notion de programme et montré le
caractère dynamique du génome (voir
Pichot)
et qui nous font replonger dans le darwinisme : |
|
Une information à disposition permettant à la cellule de s'adapter (Heams, HCC p 49-50 ) « Un des exemples désormais canoniques, bien que récemment décrit, de différenciation cellulaire reposant sur des phénomènes stochastique est l'œil de la mouche drosophile. Soit le problème suivant : un tel œil est constitué des fameuses « facettes » que l'on appelle ommatidies et qui sont au nombre de 800 par œil. Chacune de ces ommatidies est composée de huit cellules photoréceptrices. Deux d'entre elles doivent se différencier de façon à capter l'énergie lumineuse. Pour qu'un œil de drosophile fonctionne correctement, il faut que 30 % des cellules expriment un photopigment appelé rhodopsine, sensible à certaines longueurs d'onde, et que 70 % des cellules expriment une autre rhodopsine sensible à d'autres longueurs, de sorte qu'avec ces proportions, la mouche capte convenablement l'ensemble des longueurs d'onde de son spectre de vision. Imaginons qu'un programme préside à la différenciation des 800 couples de cellules concernées. Il faudrait imaginer que chaque cellule sache ce que font les autres, pour pouvoir faire le bon « choix », conduisant à la bonne proportion... Cela nécessiterait de postuler un réseau de messages spécifiques de cellule à cellule délirant ! La sélection naturelle a favorisé un mécanisme beaucoup plus économe. Grâce à un mécanisme d'équilibre bistable (que nous décrivons dans le paragraphe ci-dessous), chaque cellule a la possibilité aléatoire d'exprimer l'une ou l'autre des rhodopsines. Et la probabilité de ce choix est, pour chaque cellule de 30 %/70 %. De sorte que ce qui est une probabilité au niveau unicellulaire devient une proportion au niveau de la population. Dans ce cas de figure, plus besoin de penser un mécanisme fin de régulation, de contrôle précis de ce qui est exprimé dans chacune des cellules : c'est le hasard qui agit, de sorte que l'organe soit, macroscopiquement, fonctionnel. On peut tout à fait imaginer que les pressions évolutives aient sélectionné progressivement les individus qui permettaient de mettre en place le ratio optimal pour réaliser une bonne vision. L'expression aléatoire des cellules est donc manifestement un mécanisme de différenciation potentiellement très utile. »
«... Ce mécanisme repose
lui-même sur un fonctionnement
déterministe classique. Il suppose qu'une
molécule M peut activer deux
gènes, a ou b, aboutissant à la
synthèse respective de protéine A
ou B. De plus, A a la propriété
d'inhiber le gène b et,
réciproquement, B peut inhiber a. Ainsi,
quand un molécule M va activer un des
deux gènes, c'est celui-ci qui va prendre
le dessus en inhibant l'autre.» (Heams,
HCC) Un équilibre bistable (1) avec A et B
à la même concentration
(activité de base du gène a =
activité de base du gène b); (2)
à des concentrations différentes A
> B (activité de base de a >
activité de base de b). L'activité
d'un gène peut dépendre par exemple
de son acessibilité. En présence d'un activateur (M) qui
peut activer l'expression de l'un OU de l'autre
gène, chaque cellule finira par n'exprimer
qu'un seul gène si la repression est
suffisamment forte. Pour justifier de
l'équilibre on doit faire appel à
une rétroaction négative de chaque
protéine sur le gène qui la produit.
Je n'ai pas représenté la faible
rétroaction négative exercée
par la protéine non dominante sur son
propre gène car elle ne peut jouer un
rôle que si la quantité de
protéine est suffisante ce qui ne serait
pas le cas dans le modèle
présenté. Dans ce cas, le rapport entre les deux
protéines A et B dans la population
cellulaire dépendra de l'affinité de
M pour tel ou tel gène. Il est bien
difficile de voir ici de l'aléatoire et
l'on peine à comprendre pourquoi M ne
pourrait activer simultanément dans une
cellule les deux gènes présents. |
||||||||||||||||||||||||||||||
| |
|
8.2 - Un génome dynamique |
||||||||||||||||||||||||||||||
|
Les gènes ne sont plus juxtaposés, figés et isolés mais sont emboîtés (plusieurs produits peuvent être synthétisés à partir d'un seul gène selon l'environnement), dynamiques (le début et la fin du gène peuvent varier, le génome d'une cellule évolue avec son âge...), et interdépendants (l'expression d'un gène est sous le contrôle d'autres gènes et souvent sous le contrôle de ses produits (rétrocontrôle)...). Mais cette complexité, qui fait appel notamment à la science des réseaux, dépasse de loin le niveau du lycée. |
|
Le niveau d'expression d'un gène dépend de facteurs épigénétiques c'est-à-dire non directement liés à l'information génétique, comme par exemple le repliement de l'ADN et sa compaction autour des nucléosomes ; ces facteurs pouvant très bien être d'origine mécanique... |
|
La considération d'une information linéaire stable (gènes juxtaposés) est l'obstacle majeur à notre nouvelle compréhension du ganome. Il faut donc probablement s'endébarasser et chercher à présenters des modèles où l'ADN, inséparable des ARN et en permanente modification, n'est plus une structure filamenteuse linéaire mais est constitué de très nombreuses sous-unités, certes réunies sous une forme filamenteuse, mais non pas comme les perles d'un collier enfilées dans un ordre déterminé mais bien davantage comme les points stables d'une dynamique filamenteuse. |
|
|
||||||||||||||||||||||||||
|
|
||||||||||||||||||||||||||||||||
|
On ne peut pas se contenter d'un raisonnement simpliste 9 - Pour une génétique intelligente |
||||||
| |
|
La génétique, science des
gènes, de science de l'hérédité
(des sélectionneurs du XIX et XXème
siècles), est devenue une partie de la biologie
moléculaire avec de nombreuses applications
biotechnologiques (le génie
génétique) et des applications
médicales (ce que l'on appelle les maladies
génétiques ou les facteurs
génétiques de certaines maladies). |
||||
|
Le regard de l'enseignant n'est ni médical, ni commercial (pharmaceutique) ni technique (le niveau des techniques de la biologie moléculaire dépasse le niveau d'enseignement du lycée). Je refuse les raisonnements simplistes sur ces questions importantes qui demandent à être abordées avec prudence. |
|
Je rappelle les mots du programme : « Objectifs et mots
clés. Objectifs et
méthodes. |
|
Cet objectif de prudence a beau être présenté dans le paragraphe sur le cancer il se réfère AUSSI à celui sur la mucoviscidose.... Aucune maladie génique ne présente un déterminisme absolu comme il est imprudemment noté dans le Bordas. |
||
| |
|
9.1 - Une médecine génétique prudente : aucune maladie ne présente de déterminisme génique absolu |
||||
|
Le terme de maladie génique désigne IDÉALEMENT une maladie dont la totalité des symptômes pourrait être expliquée par l'information génétique. Elle suppose donc presque obligatoirement la soumission intellectuelle à la théorie de l'information génétique. Elle conduit à rechercher des thérapies géniques, cherchant à réparer le gène déficient ou les étapes de l'expression de l'information génétique déficientes. |
|
Historiquement Les exemples les plus classiques, et historiquement les plus anciens, concernent tous des maladies du métabolisme, pour lesquelles, il est évident que l'on peut relier assez facilement des symptômes à l'efficacité plus ou moins altérée d'une enzyme intervenant dans une réaction métabolique. Si cette idée était simple pour une bactérie ou une cellule filamenteuse de champignon, elle est beaucoup plus complexe à justifier pour un pluricellulaire, a fortiori pour l'homme. Le concept s'est développé aux alentours des années 1910 notamment grâce aux travaux d'Archibald Garrod (page sur l'alcaptonurie). |
|
Il y a aussi le cas de la drépanocytose que l'on relie à la malformation d'une unique chaîne protéique de l'hémoglobine. Mais le cas s'est compliqué du fait que chacun possède plusieurs gènes de la globine, que leur expression varie au cours de la vie et enfin, que l'on synthétise plusieurs types d'hémoglobine qui comporte 4 chaînes identiques deux à deux. Cependant, on arrive à relier de façon assez convaincante une grande majorité de symptômes à la présence de tel ou telle séquence du gène d'une chaîne de globine. Il est évident que les symptômes varient cependant chez des individus qui ont le même génotype. Son étude, la plupart du temps présentée correctement dans les manuels devrait être faite dans une perspective historique. Il existe un lien certain entre le génotype et le phénotype (au sens moderne de ces mots) mais ce lien n'est jamais absolu. |
||
| |
|
|
||||
|
Le terme maladie génétique ne signifie pas grand-chose et son sens doit être précisé. - Si un gène spécifique est suspecté d'être à l'origine de la maladie, on peut parler de maladie génique. De même si plusieurs gènes sont impliqués de façon certaine. - Si, par contre, des facteurs génétiques sont mis en évidence, ce qui est le cas pour un très grand nombre de maladies, il faut éviter d'employer le terme de génétique. On préféra donc toujours "génique", univoque, à "génétique", équivoque. |
|
Trois exemples de maladies sont détaillés dans des pages annexes : - la phénylcétonurie qui a des facteurs génétiques importants, mais qui n'est pas une maladie génique, - la mucoviscidose, qui est toujours considérée comme génique mais qui repousse nettement les limites du modèle; - la maladie d'Alzheimer qui n'est pas considérée comme une maladie génique (et qui ne l'est absolument pas), mais qui présente des facteurs génétiques, comme la plupart des maladies : un remarquable article du magazine La Recherche sur la génétique de la maladie d'Alzheimer mérite d'être cité en exemple pour son discours prudent. (Pour aller encore plus loin visitez le blog d'un médecin (Dr Martial Von Linden) qui prône une approche surtout non médicale : intéressant). |
|
Les cancers, cités dans le programme, sont des maladies très étudiées pour lesquelles plusieurs théories sont en concurrence. La piste génétique est loin d'être la plus novatrice. Je reporte son étude à une année ultérieure. Les tests génétiques qui apparaissent sur le marché (disponibles sur des sites internet basés à l'étranger) et visant au dépistage des maladies comme le diabète ou l'hypertension jouent sur la crédulité des malades, mais comment ne pas y voir une conséquence de l'erreur du déterminisme génétique, toujours enseigné sans demi-teinte. Il est bien tard pour s'inquiéter des dérives mercantiles et affirmer que "le facteur génétique n'est qu'un élément parmi d'autres", en précisant : "pour ces pathologies [qui] ne sont pas monogéniques" (mais le modèle monogénique n'est plus pertinent pour aucune maladie humaine...)... (interview de la directrice de l'Agence de Biomédecine, 16 avril 2013) |
||
| |
|
9.2 - De nombreux mécanismes génétiques peuvent être impliqués dans les mécanismes évolutifs |
||||
|
Voir fiche seconde : Qu'est-ce que l'évolution ? |
|
L'évolution est l'idée selon laquelle LES ESPÈCES se transforment au cours du temps. Il existe plusieurs théories concernant les mécanismes
de l'évolution. |
|
La sélection naturelle, que Darwin
proposa en 1859 dans son ouvrage "L'origine des
espèces", est un mécanisme dont
l'idée a été empruntée aux
sélectionneurs qui réalisent une
sélection artificielle des espèces afin
d'améliorer les caractéristiques des
espèces cultivées ou élevées. Il
l'associe avec l'idée que les espèces se
reproduisent en nombre excessif et que certains individus
doivent donc disparaître : ce seront les moins bien
adaptés; les mieux adaptés sont donc ainsi
sélectionnés par sélection naturelle.
Cette théorie de l'adaptation sera reprise
ensuite par ceux que l'on a appelés darwiniens qui
élaborent une théorie de l'évolution en
intégrant petit à petit les résultats de
la biologie des espèces (notamment de la
génétique des populations au milieu du
XXème et le mutationnisme selon lequel les
mutations sont sources de variations tant favorables que
défavorables). La théorie synthétique est
née entre 1940 et 1970 de ces développements. |
||
|
|
||||||
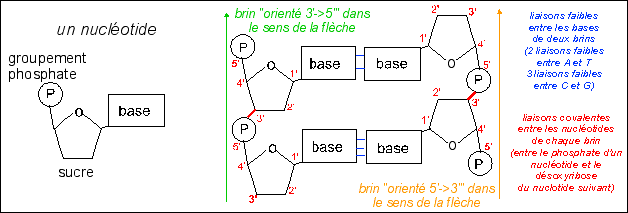
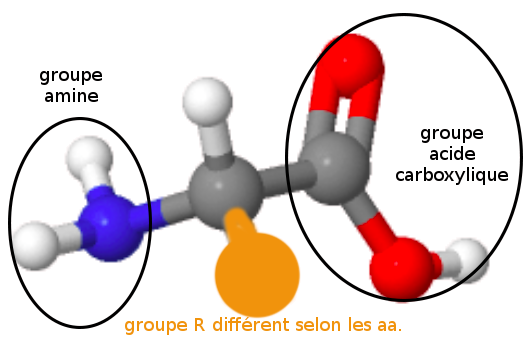
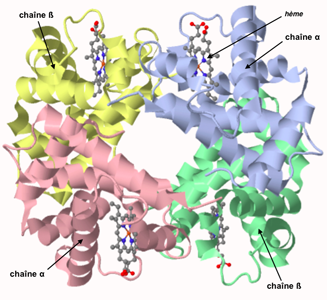

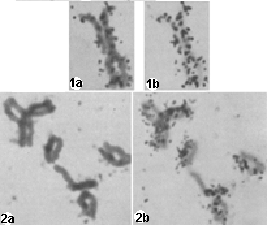 Un
aperçu
(très mauvaise qualité juste pour donner envie
d'aller voir
Un
aperçu
(très mauvaise qualité juste pour donner envie
d'aller voir 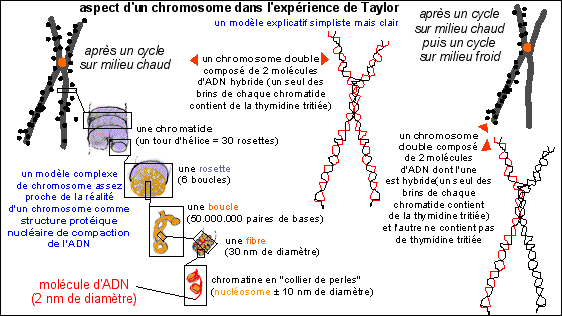
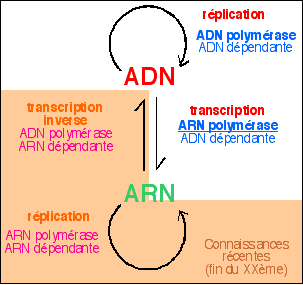

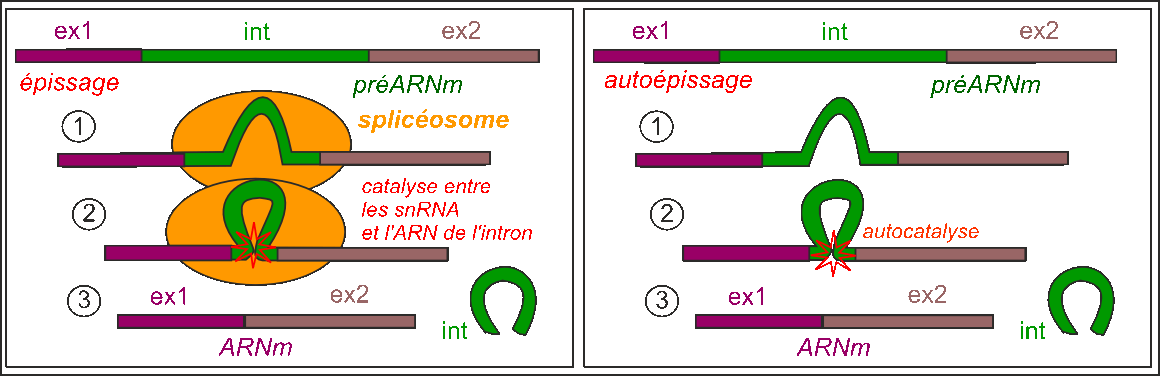
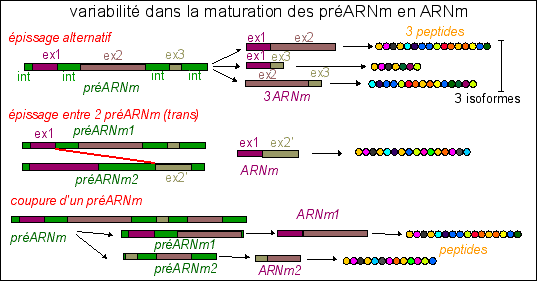 Tous les mécanismes ne sont
pas représentés, il s'agit juste d'une
illustration du cours.. De plus, les
Tous les mécanismes ne sont
pas représentés, il s'agit juste d'une
illustration du cours.. De plus, les