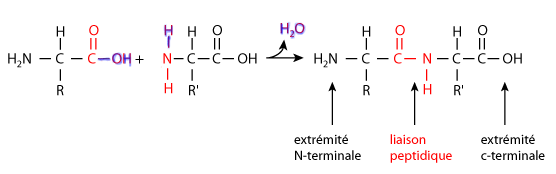
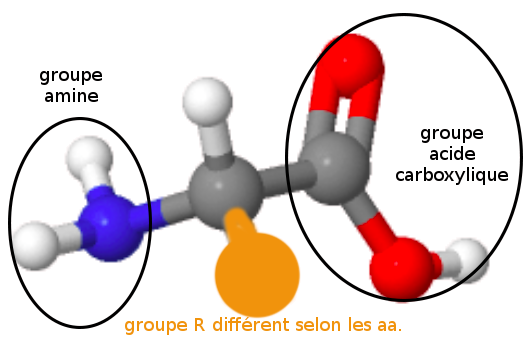
1 - Les acides aminés
Tout acide aminé (aa) est composé d'un carbone lié par covalence à un hydrogène et à 3 groupes dont un seul est variable : le groupe R.Le groupe amine (-NH2) est placé conventionnellement à gauche des représentations et le groupe acide carboxylique (-COOH) à droite. Comme le carbone central de la molécule comporte 4 groupes presque toujours différents (sauf pour la glycine où R=H), c'est un carbone asymétrique (noté C*) d'où les propriétés optiques des aa vues en chimie.
|
|
|
|
Formule
du groupement R - |
| alanine |
|
|
-CH3 |
| arginine |
|
|
-CH2-CH2-CH2-NH-C=N+H2(NH2) |
| asparagine |
|
|
-CH2-C=O(NH2) |
| acide
aspartique |
|
|
-CH2-COO- |
| cystéine |
|
|
-CH2-SH (deux
Cys peuvent s'associer en ponts disulfure
S-S) |
| glutamine |
|
|
-CH2-CH2-C=O(NH2) |
| acide
glutamique |
|
|
-CH2-CH2-COO- on dit aussi "glutamate" |
| glycine |
|
|
-H |
| histidine |
|
|
-CH2-C(NH-CH=N-)=CH avec cycle |
| isoleucine |
|
|
-CH(CH3)-CH2-CH3 |
| leucine |
|
|
-CH2-CH-CH3(CH3) |
| lysine |
|
|
-CH2-CH2-CH2-CH2-N+H3 |
| méthionine |
|
|
-CH2-CH2-S-CH3 |
| phénylalanine |
|
|
-CH2-C6H5 avec cycle benzènique |
| proline |
|
|
côté1
lié au C*-N+H2-CH2-CH2-CH2-
côté 2 lié au C* |
| sérine |
|
|
-CH2OH |
| thréonine |
|
|
-CHOH-CH3 |
| tryptophane |
|
|
-CH2-C=CH(NH)-C6H4 avec deux cycles |
| tyrosine |
|
|
-CH2-C6H4(OH) avec cycle benzènique |
| valine |
|
|
-CH-CH3(CH3) |