Michael B. Elowitz, 1,2 * Arnold J. Levine, 1 Eric D. Siggia, 2 Pierre S. Swain2
1 Laboratoire de biologie du cancer,
2 Centre d'études en physique et en biologie, Université Rockefeller, New York, NY 10021, USA.
* À qui la correspondance doit être adressée. Email: elowitm@rockefeller.edu
![]() Cette traduction mot-à-mot a pour unique but d'aider
à comprendre l'importance de cet article fondateur.
(cours
de
1èreS)
Cette traduction mot-à-mot a pour unique but d'aider
à comprendre l'importance de cet article fondateur.
(cours
de
1èreS)
Stochastic Gene Expression in a Single Cell, Michael B. Elowitz et al., Science, 297,1183 (2002)
Les populations clonales de
cellules présentent d'importantes variations
phénotypiques. Cette
hétérogénéité peut
être essentielle pour de nombreux processus
biologiques et est supposée provenir de la
stochasticité ou bruit dans l'expression des
gènes. Nous avons construit des souches
d'Escherichia coli qui permettent la détection
du bruit et la discrimination entre les deux
mécanismes par lesquels il est
généré. Les deux stochasticités,
aussi bien celle inhérente aux processus biochimiques
de l'expression des gènes (bruit intrinsèque)
que celle due aux fluctuations des autres composants
cellulaires (bruit extrinsèque) contribuent de
manière substantielle à la variation globale.
Le taux de transcription, la dynamique de régulation
et des facteurs génétiques contrôlent
l'amplitude du bruit. Ces résultats fondent
quantitativement la modélisation du bruit dans les
réseaux génétiques et
révèlent comment un petit nombre de copies de
molécules intracellulaires peut fondamentalement
limiter la précision de la régulation des
gènes.
Clonal populations of cells exhibit substantial phenotypic variation. Such heterogeneity can be essential for many biological processes and is conjectured to arise from stochasticity, or noise, in gene expression. We constructed strains of Escherichia coli that enable detection of noise and discrimination between the two mechanisms by which it is generated. Both stochasticity inherent in the biochemical process of gene expression (intrinsic noise) and fluctuations in other cellular components (extrinsic noise) contribute substantially to overall variation. Transcription rate, regulatory dynamics, and genetic factors control the amplitude of noise. These results establish a quantitative foundation for modeling noise in genetic networks and reveal how low intracellular copy numbers of molecules can fundamentally limit the precision of gene regulation.
Les cellules vivantes possèdent un tout petit nombre de copies de nombreux composants, y compris de l'ADN et des molécules régulatrices importantes (1). Aussi, les effets stochastiques dans l'expression des gènes peuvent expliquer les grandes variations observées entre cellules au sein de populations isogéniques (2, 3). Ces effets peuvent jouer un rôle crucial dans les processus biologiques, tels que le développement, en établissant des asymétries initiales qui, amplifiées par des mécanismes de rétroaction, peuvent déterminer le devenir des cellules (4). Toutefois, la mise en évidence expérimentale de la stochasticité dans l'expression des gènes est restée indirecte (5, 6). Pour tout gène spécifique, on ne sait pas si la variation entre cellules de l'abondance d'un produit est due au bruit dans l'expression du gène lui-même ou à des fluctuations dans les quantités d'autres composants cellulaires. La difficulté de distinguer expérimentalement entre ces deux possibilités a jusqu'à présent empêché la détection du bruit intrinsèque dans les cellules vivantes. L'ampleur du bruit intrinsèque lors de l'expression des gènes, et son importance par rapport à d'autres sources de variabilité entre cellules, sont des caractéristiques cellulaires fondamentales qu'il fallait mesurer.
En général, la quantité de protéine produite par un gène particulier varie entre cellules. Le bruit (défini comme l'écart type divisé par la moyenne) dans cette distribution est noté ηtot et peut être divisée en deux composantes. Puisque l'expression de chaque gène est contrôlée par les concentrations, les formes et les emplacements des molécules telles que les protéines régulatrices et les polymérases, la fluctuation de la quantité ou de l'activité de ces molécules est à l'origine des fluctuations dans l'activité du gène. Par conséquent, ils représentent des sources de bruit extrinsèques (notée ηext) qui dépendent globalement de la cellule, mais peuvent varier d'une cellule à une autre. D'autre part, si l'on considère de façon hypothétique une population de cellules identiques non seulement génétiquement, mais aussi identiques au niveau des concentrations et des états de leurs composants cellulaires, le taux d'expression d'un gène particulier pourrait encore varier d'une cellule à l'autre, du fait des événements microscopiques aléatoires qui font que telle ou telle réaction se produit et dans tel ordre. Cette stochasticité inhérente à la cellule, ou bruit intrinsèque, notée ηint, est l'autre partie du bruit total qui résulte de la nature discrète des processus biochimiques de l'expression des gènes. La précision du contrôle des niveaux de protéines régulatrices importe peu puisque le bruit intrinsèque limite fondamentalement la précision de la régulation des gènes.
D'une façon pratique, le bruit intrinsèque pour un gène donné peut être défini comme la mesure de l'écart de corrélation entre les activités de deux copies identiques de ce gène situées dans le même environnement intracellulaire (Fig. 1, A et B). Par conséquent, nous avons fabriqué deux souches repérables d'Escherichia coli en intégrant dans leur chromosome l'un des deux allèles cyan (cfp) ou jaune (yfp) de la protéine fluorescente verte. Dans chaque souche, les deux gènes rapporteurs étaient contrôlés par des promoteurs identiques. Afin d'éviter des différences systématiques dans le nombre de copies, nous avons intégré les gènes en des loci équidistants, mais sur les côtés opposés de l'origine de réplication (fig. S1). Les deux protéines fluorescentes ont montré des distributions d'intensité équivalentes et présentent donc à la fois la nécessaire indépendance et la similarité pour détecter le bruit (7).
Living cells possess very low copy numbers of many
components, including DNA and important regulatory molecules
(1). Thus, stochastic effects in gene expression may account
for the large amounts of cell-cell variation observed in
isogenic populations (2, 3). Such effects can play crucial
roles in biological processes, such as development, by
establishing initial asymmetries that, amplified by feedback
mechanisms, determine cell fates (4 ). However, experimental
evidence for stochasticity in gene expression has been
circumstantial (5, 6 ). For any particular gene, it remains
unknown whether cell-cell variation in the abundance of its
product is set by noise in expression of the gene itself or
by fluctuations in the amounts of other cellular components.
The difficulty of experimentally distinguishing between
these two possibilities has thus far precluded detection of
intrinsic noise in living cells. The magnitude of the noise
intrinsic to gene expression, and its relative importance
compared with other sources of cell-cell variability, are
fundamental characteristics of the cell that require
measurement.
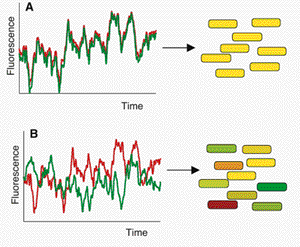
Fig. 1. Les bruits intrinsèque et extrinsèque peuvent être mesurés et séparés avec deux gènes (cfp, en vert; yfp, en rouge) commandés par des séquences régulatrices identiques. Les cellules avec la même quantité de chaque protéine apparaissent en jaune, tandis que les cellules exprimant plus d'une protéine fluorescente que l'autre apparaissent en rouge ou en vert.
(A) En l'absence de bruit intrinsèque, les deux protéines fluorescentes fluctuent de façon corrélée au fil du temps dans une seule cellule (à gauche). Alors que, dans une population, chaque cellule aura la même quantité des deux protéines, bien que ce montant sera différent d'une cellule à cause du bruit extrinsèque (à droite).
(B) L'expression des deux genes peut aussi devenir non corrélée entre cellules individuelles à cause du bruit intrinsèque (à gauche), donnant lieu à une population dans laquelle certaines cellules expriment plus d'une protéine fluorescente que l'autre.
In general, the amount of protein produced by a particular gene varies from cell to cell. The noise (defined as the standard deviation divided by the mean) in this distribution is labeled tot and can be divided into two components. Because expression of each gene is controlled by the concentrations, states, and locations of molecules such as regulatory proteins and polymerases, fluctuations in the amount or activity of these molecules cause corresponding fluctuations in the output of the gene. Therefore, they represent sources of extrinsic noise (denoted ηext) that are global to a single cell but vary from one cell to another. On the other hand, consider a population of cells identical not just genetically but also in the concentrations and states of their cellular components. Even in such a (hypothetical) population, the rate of expression of a particular gene would still vary from cell to cell because of the random microscopic events that govern which reactions occur and in what order. This inherent stochasticity, or intrinsic noise, denoted ηint is that remaining part of the total noise arising from the discrete nature of the biochemical process of gene expression. No matter how accurately the levels of regulatory proteins are controlled, intrinsic noise fundamentally limits the precision of gene regulation.
Operationally, intrinsic noise for a given gene may be
defined as the extent to which the activities of two
identical copies of that gene, in the same intracellular
environment, fail to correlate (Fig. 1, A and B). Therefore,
we built strains of Escherichia coli, incorporating the
distinguishable cyan (cfp) and yellow (yfp) alleles of green
fluorescent protein in the chromosome. In each strain, the
two reporter genes were controlled by identical promoters.
To avoid systematic differences in copy number, we
integrated the genes at loci equidistant from, and on
opposite sides of, the origin of replication (fig. S1). The
two fluorescent proteins exhibited statistically equivalent
intensity distributions and thus displayed the necessary
independence and equivalence to detect noise (7 ).
Fig. 1. Intrinsic and extrinsic noise can be measured and distinguished with two genes (cfp, shown in green; yfp, shown in red) controlled by identical regulatory sequences. Cells with the same amount of each protein appear yellow, whereas cells expressing more of one fluorescent protein than the other appear red or green. (A) In the absence of intrinsic noise, the two fluorescent proteins fluctuate in a correlated fashion over time in a single cell (left). Thus, in a population, each cell will have the same amount of both proteins, although that amount will differ from cell to cell because of extrinsic noise (right). (B) Expression of the two genes may become uncorrelated in individual cells because of intrinsic noise (left), giving rise to a population in which some cells express more of one fluorescent protein than the other.
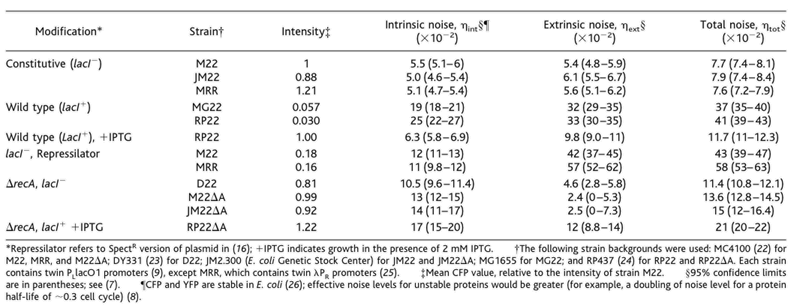
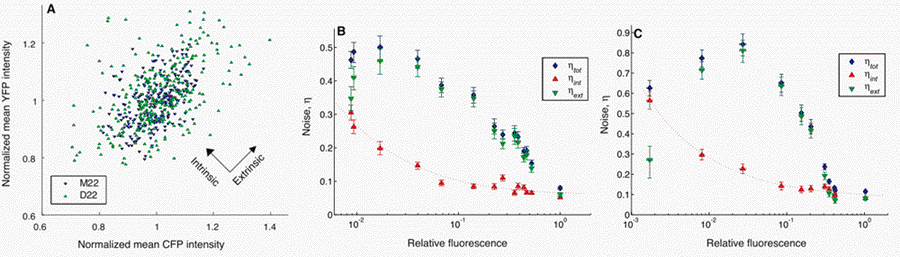
Pour les mesures, les cellules ont été cultivées dans du milieu LB et photographiés à travers des jeux de filtres fluorescents cfp et yfp ainsi qu'en contraste de phase (Fig. 2) (7). Un système d'analyse d'image informatisée a identifié les cellules et quantifié leur intensité moyenne de fluorescence. Les bruits intrinsèque et extrinsèque peuvent être déterminés à partir de la représentation graphique de la CFP en fonction de l'intensité de fluorescence YFP dans chaque cellule (Fig. 3A) (7). La valeur de ηint indique la différence relative moyenne de l'intensité de fluorescence des deux protéines rapporteuses dans la même cellule; par exemple, si ηint = 0,25, alors les deux couleurs diffèrent généralement d'environ 25%. Étant donné que ηint et ηext ont des contributions orthogonales au bruit total, ηtot, les trois valeurs du bruit satisfont à la relation ηint2+ ηext2 = ηtot2 (7, 8). Les mesures de ces variables pour les différentes souches et conditions sont présentées dans le Tableau 1.
Pour déterminer l'importance du bruit in vivo, nous avons commencé avec les conditions d'expression de gènes les moins bruyantes obtenues sans feed-back: ceux de promoteurs de base résistants qui gouvernent l'expression de protéines stables. Plus précisément, nous avons construit des souches incorporant des promoteurs artificiels lac-repressibles (9) au sein de souches lac-, dans lesquelles le gène répresseur lac, ladI, est supprimé. Nous avons obtenu de faibles niveaux de bruit (ηint ≈ 0.05) et une faible variation globale entre les cellules (ηtot ≈ 0,08) dans ces souches (Fig. 2E). Nous avons obtenu des résultats similaires dans une autre souche incorporant deux copies du promoteur un peu plus résistant λPR (tableau 1). Ces résultats indiquent (i) que l'expression du gène constitutif peut être remarquablement uniforme, sous certaines conditions, et (ii) que cet état à faible bruit ne dépend pas strictement de la séquence d'un promoteur particulier.
Peu de gènes naturels d'E. coli sont transcrits aussi intensément que ceux des promoteurs dérivés de phages (10). Pour voir la quantité de bruit il y a à de faibles taux de transcription, nous avons déplacé les rapporteurs chez plusieurs souches d' E. coli de type sauvage (lacI*), où ne sont produits que de 3 à 6% de protéines. Dans ces conditions, le bruit intrinsèque et extrinsèque sont tous deux augmentés d'un facteur de ~ 5 (Fig. 2, A et D, et tableau 1). L'effet est réversible : l'addition d'isopropyl β-D-thiogalactopyranoside (IPTG) à saturation, qui se lie au répresseur lac et l'inactive, restaure approximativement le bruit (à la fois ηint et ηext) et l'expression de la protéine fluorescente à leurs valeurs dans les souches lac- (Fig. 2B). Ainsi, l'augmentation du bruit observée dans les souches sauvages peut être attribuée directement à l'activité de lacI et à la réduction correspondante du taux de transcription. lacI affecte aussi le bruit extrinsèque en l'augmentant par un facteur de ~5 à ~ 0,3. Ce changement indiquant la présence d'une variation de l'expression lacI entre les cellules (8, 11).
For measurement, cells
were grown in LB medium and photographed through cfp and yfp
fluorescence filter sets and in phase contrast (Fig. 2) (7
). A computerized image analysis system identified cells and
quantified their mean fluorescent intensities. Both
intrinsic and extrinsic noise could be determined from plots
of CFP versus YFP fluorescence intensity in individual cells
(Fig. 3A) (7 ). The value of int indicates the mean relative
difference in fluorescence intensity of the two reporter
proteins in the same cell; for instance, if int 0.25, then
the two colors typically differ by about 25%. Because int
and ext make orthogonal contributions to the total noise,
tot, the three noise values satisfy the relation int2 ext2
tot2 (7, 8). Measurements of these variables for various
strains and conditions are presented in Table 1.
To determine the importance of noise in vivo, we began with
the least noisy gene expression conditions obtainable
without feedback: strong constitutive promoters driving the
expression of stable proteins. Specifically, we constructed
strains incorporating artificial lacrepressible promoters
(9) in lac&endash; strain backgrounds, in which the lac
repressor gene, lacI, is deleted. We obtained low noise
levels ( int 0.05) and low cell-cell variation overall ( tot
0.08) in these strains (Fig. 2E). We obtained similar
results in another strain incorporating two copies of the
somewhat stronger promoter P R ( Table 1). These results
indicate (i) that constitutive gene expression can be
remarkably uniform under some conditions, and (ii) that this
low noise state does not strictly depend on a particular
promoter sequence.
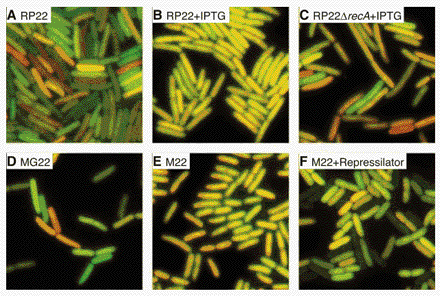
Fig. 2. Bruit dans des images de fluorescence CFP et YFP chez E. coli. combinés dans les canaux vert et rouge, respectivement.
(A) Dans la souche RP22, avec les promoteurs réprimés par le gène de type sauvage lacI, le rouge et le vert indiquent des quantités importantes de bruit intrinsèque.
(B) RP22 cultivée en présence d'un inducteur lac, 2 mM d'IPTG. Les deux protéines fluorescentes sont exprimées à des niveaux plus élevés et les cellules présentent moins de bruit.
(C) comme en (B), à l'exception du gène recA supprimé, ce qui augmente le bruit intrinsèque.
(D) une autre souche de type sauvage, MG22, présente des caractéristiques similaires à celles du bruit de RP22.
(E) Les niveaux d'expression et le bruit de la souche M22 avec lacI non réprimé sont similaires à ceux des souches lacI induites par l'IPTG (B).
(F) des cellules M22 régulées par la Repressilator (16), un réseau oscillant qui amplifie le bruit intrinsèque.
Few natural E. coli genes are transcribed as strongly as these phage-derived promoters (10). To see how much noise there is at lower rates of transcription, we moved the reporters into several wild-type (lacI ) E. coli strains, where they produced only 3 to 6% as much protein. Under these conditions, both intrinsic and extrinsic noise increased by a factor of 5 (Fig. 2, A and D, and Table 1). The effect was reversible: Addition of saturating amounts of isopropyl -D-thiogalactopyranoside (IPTG), which binds and inactivates the lac repressor, restored noise (both int and ext) and amounts of fluorescent protein expression to their approximate values in lac&endash; strains (Fig. 2B). Thus, the noise increase found in wild-type strains can be attributed directly to the activity of lacI and the corresponding reduction of transcription rate. lacI affects extrinsic noise as well, increasing it by a factor of 5, to 0.3. This change indicates the presence of cell-cell variation in lacI expression (8, 11).
Fig. 2. Noise in E. coli. CFP and YFP fluorescence images were combined in the green and red channels, respectively. (A) In strain RP22, with promoters repressed by the wild-type lacI gene, red and green indicate significant amounts of intrinsic noise. (B) RP22 grown in the presence of lac inducer, 2 mM IPTG. Both fluorescent proteins are expressed at higher levels and the cells exhibit less noise. (C) As in (B), except the recA gene has been deleted, increasing intrinsic noise. (D) Another wild-type strain, MG22, shows noise characteristics similar to those of RP22. (E) Expression levels and noise in unrepressed lacI strain M22 are similar to those in lacI strains induced with IPTG (B). (F) M22 cells regulated by the Repressilator (16), an oscillatory network that amplifies intrinsic noise.
Les modèles d'expression stochastique des gènes prévoient que le bruit intrinsèque augmente lorsque la quantité de transcrits diminue (8, 12). Pour réprimer plus efficacement les gènes rapporteurs, nous avons introduit un plasmide exprimant de manière constitutive le répresseur lac (7) dans les souches délétées d'autre part pour lacI. Nous avons ajouté différentes quantités d'IPTG aux cultures en croissance (Fig. 3, B et C). Le bruit intrinsèque est beaucoup plus important en présence du plasmide lacI à cause d'un taux de transcription réduit, mais il a diminué sensiblement lorsque l'IPTG a été ajouté. ηint devrait diminuer selon ηint2 ≈ (c1/ m) + c2 , où m est l'intensité de fluorescence de la cellule (supposée être proportionnelle à la moyenne du nombre de transcrits), et c1 et c2 sont des constantes données par les paramètres microscopiques (7). Cette formule rend compte des données, la souche D22 présentant un bruit intrinsèque plus élevé que M22 à tous les niveaux d'expression (fig. 3, B et C).
Le bruit extrinsèque, ηext, se comporte très différemment en fonction de la concentration en IPTG. Alors que ηint décroît de façon monotone, ηext atteint un maximum à des taux intermédiaires de transcription. En conséquence, la variabilité totale entre cellules (ηtot) ne détermine pas seule le bruit intrinsèque. La présence d'un maximum pour ηext peut s'expliquer par la variation entre cellules de la concentration de lacI (13). Fait intéressant, ηext est sensiblement plus faible dans les cellules portant une copie chromosomique de lacI que dans les cellules portant une copie plasmidique du gène (à des niveaux d'expression comparables, voir le tableau 1 et la Fig. 3). Ceci est cohérent avec une plus grande variabilité du nombre de copies du gène pour le gène lacI d'origine plasmidique que pour sa version de type sauvage chromosomique (14). Les bruits extrinsèque et intrinsèque doivent être combinés pour expliquer les variations observées ; les mécanismes de régulation, comme la rétroaction négative, visant surtout à supprimer le bruit (15), doivent ainsi tenir compte des deux sources de bruit.
Models of stochastic gene expression predict that intrinsic noise should increase as the amount of transcript decreases (8, 12). To more effectively repress the reporter genes, we introduced a plasmid constitutively expressing the lac repressor (7 ) into strains otherwise deleted for lacI. We added different amounts of IPTG to growing cultures (Fig. 3, B and C). Intrinsic noise was much larger in the presence of the lacI plasmid because of reduced transcription rate, but it fell substantially as IPTG was added. int is expected to decrease as int2 (c 1/m) c2, where m is the fluorescence intensity of the cell (assumed to be proportional to the average number of transcripts), and c 1 and c2 are constants given by the microscopic parameters (7 ). This form fits the data, with strain D22 exhibiting higher amounts of intrinsic noise than M22 at all levels of expression (Fig. 3, B and C).
The extrinsic noise,
ext, behaves very differently as a function of IPTG
concentration. Whereas int decreases monotonically, ext
displays a maximum at intermediate rates of transcription.
As a result, total cell-cell variability ( tot) does not
uniquely determine intrinsic noise. The presence of a
maximum in ext may be explained as a result of cell-cell
variation in the concentration of lacI (13).
Interestingly, ext is substantially smaller in cells
carrying a chromosomal copy of lacI than it is in
cells carrying a plasmid-borne copy of the gene (at
comparable expression levels; see Table 1 and Fig. 3). This
is consistent with greater variability in copy number for
the plasmid-borne lacI gene than for its wild-type
chromosomal version (14 ). Extrinsic and intrinsic noise
must be combined to explain the observed amount of
variation; regulatory mechanisms aimed at suppressing noise,
such as negative feedback (15), need to respond to both
sources.
Table 1. Measurements of noise in selected strains.
Tableau 1. Les mesures de bruit dans une sélection de souches.
 |
||
|
Fig. 3.
Quantification de bruit. |
Fig. 3. Quantification of noise. (A) Plot of fluorescence in two strains: one quiet (M22) and one noisy (D22). Each point represents the mean fluorescence intensities from one cell. Spread of points perpendicular to the diagonal line on which CFP and YFP intensities are equal corresponds to intrinsic noise, whereas spread parallel to this line is increased by extrinsic noise. (B) Noise versus rate of transcription in strain M22 (recA , lacI&endash;), with lacI supplied by plasmid pREP4 (7). Fluorescence levels (x axis) are population means. The right most point represents the strain without pREP4 and therefore is fully induced; its value, set to 1.0, was used to normalize all fluorescence intensities. IPTG (0 to 2 mM) was added to cultures and ηtot, ηint, and ηext were measured. Error bars are 95% confidence intervals. Dashed line fits int2 (c1/m) c2, where m fluorescence intensity (x axis), c 1 7 10 4, and c2 3 10 3. (C) Noise versus induction level in recA&endash;lacI&endash; strain D22, containing plasmid pREP4. All notations are as in (B). In the fit, c 1 5 10 4 and c2 1 10 2. |
|
Pour modéliser le comportement des circuits de régulation de la transcription, il est essentiel de comprendre l'interaction entre les dynamiques de régulation et le bruit. Nous avons instauré un réseau synthétique oscillatoire, appelé Repressilator (7, 16), dans la souche M22 (tableau 1a et Fig. 2F). Ce réseau conduit à la synthèse périodique de lacI, qui induit l'activation/désactivation répétitive de deux promoteurs. De grandes variations de l'intensité de fluorescence globale se produisent (ηtot élevé). Une autre conséquence est une augmentation significative de ηint (par rapport à celui d'une souche similaire avec le même taux moyen de la transcription, voir le tableau 1). Ceci est cohérent avec les prédictions théoriques que le bruit plus élevé au cours de la mise en place qu'une fois l'état d'équilibre atteint (17). Ainsi, une dynamique de régulation peut entraîner des changements substantiels dans le niveau de bruit.
Si le niveau de bruit dans une cellule est génétiquement déterminé, alors des souches différentes doivent présenter différents niveaux basaux de bruit (intrinsèques et extrinsèques). Par conséquent, nous avons inséré les deux gènes rapporteurs dans différents environnements génétiques.
La quantité de bruit était similaire dans la plupart des souches, mais pour l'une, D22, le niveau était environ deux fois plus élevé (Fig. 3, B et C, et le tableau 1). Le génotype connu de cette souche diffère des autres qui sont moins bruyants par la délétion du gène recA, ce qui suggère que le manque de RecA est responsable de l'augmentation du bruit. En accord avec cette hypothèse, la transduction de l'allèle recA dans des souches moins bruyantes telles que M22, JM22 et RP22 a été suffisante pour augmenter substantiellement ηint (Fig. 2, B et C, et le tableau 1). Cette augmentation du bruit dans les cellules recA ne dépend pas d'une perte de viabilité (18). RecA agit sur les fourches de réplication bloquées (19), ce qui suggère que l'augmentation du bruit peut provenir de différences transitoires dans le nombre de copies de différentes parties du chromosome.
Ces résultats montrent que les types de bruits intrinsèques et extrinsèques sont importants dans la variation de l'expression des gènes entre cellules. Les deux types de bruit semblent également apparaître dans toutes les réactions intracellulaires impliquant un petit nombre de réactifs. Tout composant cellulaire qui présente des fluctuations intrinsèques de sa propre concentration agira comme une source de bruit extrinsèque pour d'autres composants avec lesquels il interagit. Ainsi, compte tenu du bruit substantiel mesuré ici, le fonctionnement fiable de la cellule peut nécessiter des réseaux génétiques qui suppriment les fluctuations ou leur résistent (15, 20). En même temps, il est clair que le bruit, s'il est amplifié, offre la possibilité de générer une hétérogénéité à long terme dans une population clonale.
To be able to model the behavior of transcriptional regulatory circuits, it is essential to understand the interplay between regulatory dynamics and noise. We introduced a synthetic oscillatory network, termed the Repressilator (7, 16 ), into strain M22 ( Table 1 and Fig. 2F). This network causes periodic synthesis of lacI, which repeatedly turns both promoters on and off. Large excursions in overall fluorescence intensity occur (high tot). An additional consequence is significant increases in int (compared with that in a similar strain with the same mean rate of transcription; see Table 1). This is consistent with theoretical predictions that noise is greater during the approach to, rather than at, a steady state (17 ). Thus, regulatory dynamics can cause substantial changes in noise levels.
If the amount of noisiness in a cell is genetically determined, then different strains might exhibit different basal noise levels (both intrinsic and extrinsic). Therefore, we inserted the two reporter genes in various genetic backgrounds. The amount of noise was similar in most strains, but one, D22, displayed about twice the amount of noise (Fig. 3, B and C, and Table 1). The known genotype of this strain differed from that of a related, less noisy, strain only by deletion of the recA gene, which suggests that lack of RecA was responsible for the increased noise.
In agreement with this hypothesis, transduction of the recA allele into less noisy strains such as M22, JM22, and RP22 was sufficient to substantially increase int (Fig. 2, B and C, and Table 1). This increased noise in recA cells does not depend on a loss of viability (18). RecA acts to rescue stalled replication forks (19), which suggests that increased noise may arise from transient copynumber differences between different parts of the chromosome.
These results show that intrinsic and extrinsic classes of noise are important in setting cell-cell variation in gene expression. Both types of noise should similarly occur in all other intracellular reactions involving small numbers of reactants. Any cellular component that suffers intrinsic fluctuations in its own concentration will act as a source of extrinsic noise for other components with which it interacts. Thus, given the substantial noise measured here, reliable functioning of the cell may require genetic networks that suppress, or are robust to, fluctuations (15, 20). At the same time, it is clear that noise, if amplified, offers the opportunity to generate long-term heterogeneity in a clonal population.
References and Notes
1. P. Guptasarma,
Bioessays 17, 987 (1995).
2. J. L. Spudich et al., Nature 262, 467 (1976).
3. H. H. McAdams et al., Proc. Natl. Acad. Sci. U.S.A. 94,
814 (1997).
4. P. Heitzler et al., Cell 64, 1083 (1991).
5. M. S. Ko, Bioessays 14, 341 (1992).
6. S. Fiering et al., Bioessays 22, 381 (2000).
7. See supporting data on Science Online.
8. P. S. Swain et al., Proc. Natl. Acad. Sci. U.S.A., in
press.
9. R. Lutz et al., Nucleic Acids Res. 25, 1203 (1997).
10. U. Deuschle et al., EMBO J. 5, 2987 (1986).
11. P. C. Maloney, B. Rotman, J. Mol. Biol. 73, 77
(1973).
12. J. Paulsson, M. Ehrenberg, Q. Rev. Biophys. 34, 1
(2001).
13. At high IPTG concentrations, cells are fully induced,
regardless of the amount of lacI they contain,
whereas at low IPTG concentrations they are uniformly
repressed. In both cases, lacI fluctuations are
buffered (by excess IPTG or excess lacI,
respectively), and hence ηext must be small.
However, between these two extremes, fluctuations in the
amount of lacI cause corresponding fluctuations in
the transcription rate of the reporter genes, and thus ηext
reaches a maximum value. See (8) for a more detailed
treatment.
14. D. Boyd, et al., J. Bacteriol. 182, 842 (2000).
15. A. Becskei, L. Serrano, Nature 405, 590 (2000).
16. M. B. Elowitz, S. Leibler, Nature 403, 335 (2000).
17. M. Thattai, A. van Oudenaarden, Proc. Natl. Acad. Sci.
U.S.A. 98, 8614 (2001).
18. Deletion of recA has pleiotropic effects, including an
increased fraction of nonviable cells in growing cultures
(21). By following microcolony formation with time-lapse
microscopy (7), we checked whether the observed noise
increase in be attributed to a subpopulation of nonviable
cells. All M22 (recAnies (10 of 10), whereas some (2 of 14)
of the M22 viable cells at the initial timepoint were
consistent with those previously obtained with larger
samples.
19. M. M. Cox et al.,
Nature 404, 37 (2000).
20. U. Alon et al., Nature 397, 168 (1999).
21. F. N. Capaldo, S. D. Barbour, Basic Life Sci. 5A, 405
(1975).
22. M. J. Casadaban, J. Mol. Biol. 104, 541 (1976).
23. D. Yu et al., Proc. Natl. Acad. Sci. U.S.A. 97, 5978
(2000).
24. J. S. Parkinson, S. E. Houts, J. Bacteriol. 151, 106
(1982).
25. B. J. Meyer, R. Maurer, M. Ptashne, J. Mol. Biol. 139,
163 (1980).
26. J. B. Andersen et al., Appl. Environ. Microbiol. 64,
2240 (1998).
27. We thank U. Alon, S. Bekiranov, J. Dworkin, D. Endy, C.
Guet, R. Kishony, S. Leibler, D. O'Carroll, N. Rajewsky, B.
Shraiman, D. Thaler, and especially M. G. Surette for
conversations and suggestions; A. Teresky and the Levine Lab
for help; and J. Paulsson for his suggestion about the
extrinsic noise profile. Recombination-proficient strains
were provided by D. Court.
Supported by the Burroughs-Wellcome Fund and the Seaver Institute (M.B.E.) and by NIH grant GM59018 (P.S.S.).
Eléments
supplémentaires en ligne
(ci-dessous)
www.sciencemag.org/cgi/content/full/297/5584/1183/ DC1
Materials and Methods
Fig. S1
14 Février 2002; accepté lr 13 June 2002
Matériels et méthodes
Souches et plasmides. Les
plasmides exprimant CFP ou YFP (codons de type sauvage
codons, développés par l'Université de
Washington Yeast Resource Center) à partir de
plusieurs promoteurs, y compris PLlacO1
(S1) et &lambad;PR (S2) ont
été construits. Le gène de
résistance aux antibiotiques, le promoteur, le site
de liaison du ribosome, le gène de la protéine
fluorescente, et les terminateurs de transcription de ces
plasmides ont été amplifiés par PCR et
intégrés à des loci chromosomiques
particuliers par recombinaison homologue (S3), en
utilisant des amorces de queue de 50 pb spécifiant la
région cible. Pour le locus galK, les amorces
suivantes, dérivées de celles décrites
précédemment (S3), ont
été utilisées pour amplifier les
unités de transcription de cfp (les homologies
pour le chromosome de la bactérie E. coli sont
indiquées en lettres majuscules; les segments
homologues au plasmide modèle sont en minuscules) :
TTCATATTGTTCAGCGACAGCTTGCTGTACGGCAGGCACCAGCTCTTCCG
cacgttaagggattttggtca et
GTTTGCGCGCAGTCAGCGATATCCATTTTCGCGAATCCGGAGTGTAAGAA
cgcctttgagtgagctgata. Pour l'intégration dans le
locus intC, les amorces suivantes ont
été utilisées pour amplifier yfp
:
ATAGTTGTTAAGGTCGCTCACTCCACCTTCTCATCAAGCCAGTCCGCCCA
tgaagtcagccccatacgat et
CCGTAGATTTACAGTTCGTCATGGTTCGCTTCAGATCGTTGACAGCCGCA
gagtcagtgagcgaggaagc.
Les inserts chromosomiques ont
été déplacés vers d'autres
souches, le cas échéant, par transduction P1
(S4). En outre, pour la répression des
gènes rapporteurs (comme sur la Fig. 3, B et C), le
plasmide pREP4 (Qiagen) a été introduit dans
les cellules. pREP4 porte la mutation
lacIq du promoteur pour un
haut niveau d'expression du répresseur lac.
Pour les expériences avec le Repressilator, nous
avons remplacé le gène ampR
du plasmide original (S5) avec une cassette de
résistance à la spectinomycine (S1).
Équivalence statistique de
gènes rapporteurs.
Nos méthodes supposent que les deux gènes
rapporteurs soient régulés de façon
identique, en moyenne. En d'autres termes, nous supposons
que les distributions des quantités de CFP et YFP par
cellule sont les mêmes. Pour vérifier cette
hypothèse, des distributions de souches d'une seule
couleur contenant des paires promoteurs
lac-répressibles (M22 et D22, tableau 1) ont
été mesurées et soumis à un test
de Kolmogorov-Smirnov. Dans les deux cas, les distributions
individuelles normalisées pour la CFP et l'YFP
étaient compatibles avec l'hypothèse
nulle, selon laquelle ils ont
tous deux été échantillonnés
avec la même distribution sous-jacente (P = 0,71 et
0,68 respectivement).
Pour vérifier les interactions possibles entre PCP et YFP, nous avons construit des souches contenant un promoteur constitutif &lambad;PR entraînant une couleur (PCP ou YFP), et le promoteur lac-inductible (PLlacO1) entraînant l'autre. Aucune différence reproductible en protéine constitutive n'a été trouvée parmi les distributions mesurées avec différentes quantités d'IPTG. Avec la souche contruite avec le promoteur lac-inductible, les quantités moyennes des deux protéines peuvent varier d'une façon co-linéaire sur deux ordres de grandeur.
Protocole de mesure. Les cultures ont été menées pendant une nuit dans du milieu LB à 32°C. Le lendemain, 2 mL de LB frais a été inoculé avec 20 µL de la culture de la nuit, cultivé à 32°C à OD600= 0,2 à 0,3, et transféré dans la glace. ~2 µL des cellules ont été placées entre lame et lamelle avec 1,5% d'agarose dans du PBS. Environ 10 champs ont été acquis à un grossissement de 100X avec un microscope à fluorescence Leica DMIRB / E, une caméra CCD (Orca II, Hamamatsu) refroidie et le logiciel courant de contrôle du microscope. Pour chaque champ, des images en contraste de phase et en fluorescence (cube CFP = Chroma, # 31044v2, et le cube YFP = Chroma, # 41028) ont été acquises. L'analyse a été faite hors ligne avec le logiciel courant. Les images de fluorescence ont été corrigées pour un éclairage homogène. Le programme a identifié des cellules individuelles sur des images à contraste de phase (les formes ont été filtrées pour éliminer les zones sombres sans cellules ou des amas de cellules), et extrait les valeurs des pixels des régions correspondantes des images de fluorescence.
Comme témoin négatif, les cellules exprimant la GFP ont été mesurés à travers les filtres CFP, YFP, et la GFP (Chroma, # HQ41001) et traitées comme ci-dessus. La résultante du «bruit», ηintO ≈ 0.02 se rapproche du niveau d'erreur du système de mesure dans son ensemble. Les valeurs du bruit Intrinsèque peuvent donc surestimer les vraies valeurs par un facteur de &radic:[1+ η2intO / η2intO] (¾ 12% pour l'ensemble des données).
Définitions du bruit et paramétrage des données. Le bruit intrinsèque (ηint) , le bruit extrinsèque (ηext) and la variation totale (ηtot) ont été définis comme suit :
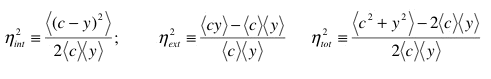
Ici le ième élement des vecteurs c et y contient la moyenne de l'intensité CFP ou YFP respectivcement de la ième cellule de l'échantillon. Les parenthèses angulaires représentent des moyennes sur la population cellulaire. Ces définitions considèrent les données cfp et yfp comme symétriques, permettant à ηint d'être interprété comme la distance normalisée r.m.s. de la droite définie par CFP=YFP (Fig.2B), et satisfaisant à ηint2+ ηext2 = ηtot2 (S6). Ainsi, lorsque l'une des deux sources de bruit diminue à 0, l'autre définit le entièrement le bruit total. Les barres d'erreur (voir figure 3) ont été calculées par la méthode du bootstrap à partir des distributions réelles.
Dans la forme ηint2 ≈ (c1/ m) + c2 , le terme c1 représente l'augmentation du bruit intrinsèque qui accompagne une diminution du taux de transcription. c2 devrait être non nul en raison des fluctuations de variables extrinsèques (S6). En outre, les différences stochastiques dans le temps de réplication peuvent également intervenir ; en particulier, notez la valeur accrue de c2 dans les cellules ΔrecA (Fig. 3, B et C).
Microcolonies. Les microcolonies ont été cultivées sur de l'agarose Seaplaque à faible point de fusion (BMA) dans milieu minimum M9 complété avec du glycérol 0,2%, des acides casaminés 0,01%, de la biotine (0,15 µg/mL), et de la thiamine 1,5 µM. Les cellules ont été préparées comme ci-dessus, mais avec une densité au vingtième. Des logiciels personnalisés et ImageProPlus (Media Cybernetics) ont été utilisés pour contrôler les fonctions automatisées du microscope et maintenir la netteté pendant l'acquisition.
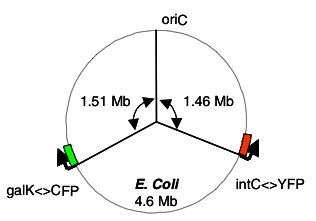
Figure
complémentaire Références et
notes S1. R. Lutz, H. Bujard,
Nucleic Acids Res. 25, 1203
(1997). Fig. S1. Une carte du
chromosome d'E. coli avec l'origine de
réplication (oriC), et les loci
cfp et yfp indiqués. Les
emplacements ont été choisis pour
éviter systématiquement les
différences dans le nombre de copies du
gène, mais en restant sensible aux
variations stochastiques,. Le symbole <>
montre remplacement par recombinaison homologue
(S3).
S2. BJ Meyer, R. Maurer, M. Ptashne, J. Mol.
Biol. 139, 163 (1980).
S3. D. Yu et al., Proc. Natl. Acad. Sci. USA
97, 5978 (2000).
S4. JH Miller, Short Course in Bacterial
Genetics: A Laboratory Manual A Laboratory Manual
and Handbook for Escherichia Coli and
Related Bacteria (Cold Spring Harbor Laboratory
Press, Cold Spring Harbor, New York, 1992).
S5. MB Elowitz, S. Leibler, Nature
403, 335 (2000).
S6. PS Swain, MB Elowitz, ED Siggia, en
préparation.