|
retour cours
de seconde, cours
de 1èreS
|
|
|
|
|
|
les* ADN : acides
désoxyribonucléiques
|
|
|
|
* il existe plusieurs
conformations possibles et donc plusieurs types de
molécules d'ADN; on parle cependant de l'ADN comme
un type de molécule unique.
|
|
Les acides nucléiques sont de longues chaînes de
nucléotides.
Les nucléotides d'une même chaîne sont
associés par des liaisons fortes.
|
|
L'ADN est une double chaîne de nucléotides
(c'est une molécule bicaténaire; on dit aussi
qu'elle a 2 brins) enroulée en hélice.
|
|
Les nucléotides de l'ADN contiennent du
désoxyribose et 4 bases azotées (A,T, C, G).
Les deux chaînes sont associées par des liaisons
faibles entre bases complémentaires (A =T, C =
G).
|
|
nucléotide
(désoxyribonucléotide)
|
|
|
nucléoside
(désoxyribonucléoside)
|
+
|
groupement phosphate
|
|
|
base (azotée)
|
+
|
sucre
|
|
A
|
adénine
|
bases puriques
|
désoxyribose
C5H10O4
|
PO43-
(en solution aqueuse, c'est
l'acide phosphorique: H3 PO4)
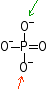
|
|
G
|
guanine
|
|
C
|
cytosine
|
bases pyrimidiques

|
|
T
|
thymine
|
|
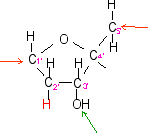
la flèche
rouge indique
le point de liaison avec le sucre
du nucléotide auquel appartient la base
|
les flèches
rouges indiquent les
liaisons avec la base à gauche et le
groupement phosphate à droite
la flèche
verte indique le point
de liaison éventuel (en 3') avec le
groupement phosphate d'un autre
nucléotide
|
la flèche
rouge indique le point de
liaison avec le sucre (en 5') du
nucléotide auquel appartient le
groupement phosphate
la flèche
verte indique la liaison
éventuelle avec le sucre d'un autre
nucléotide
|
|
|
* cliquez sur le nom des bases pour afficher
leur formule développée sous forme d'une
petite image dans une nouvelle page.
|
|
** les carbones du désoxyribose sont
numérotés de 1' à 5' (les n°s sans
les ' sont ceux des carbones et des azotes de la base
azotée)
|
|
*** le désoxyribose, comme son nom
l'indique, a perdu un oxygène (en 2') par rapport au
ribose.
|
|
|
|
|
Avertissement: les
logiciels dit de "Visualisation" des molécules
en 3D sont plutôt des logiciels de Simulation;
je parlerai de Simualisation.
|
|
|
Pour simualiser les molécules
l'applet java Jmol (http://jmol.sourceforge.net/)
est probablement le moyen le plus accessible sur tous les
matériels (pour système Windows, Linux ou OS -
tous les formats de fichiers moléculaires sont
acceptés). Cette applet est en OPEN SOURCE !!!!
(c'est à dire non seulement gratuite mais disponible
pour que tous puissent améliorer le code et les
fonctions du logiciel).
|
|
Attention la version 10 complète avec
les doc et le source pèse 22,7 Mo mais cela vaut le
coup de passer deux heures (à 56Ko/s) à la
charger... (Pour démarrer lancer jmol.jar). On peut
aussi utiliser les mêmes fonctionnalités que
l'applet (JmolApplet.jar) dans des pages html en
utilisant javascript (bibliothèque jmol.js),
ce qui a été fait dans cette page.
|
|
Pour se procurer des fichiers de
molécules il faut plus ou moins comprendre l'anglais,
même si quelques molécules sont disponibles sur
le site de l'inrp (http://www.inrp.fr/Acces/Biogeo/model3d/data3d.htm),
pas toujours dans un format utilisable sur un Mac (.exe) !!!
ou mieux sur le site http://librairiedemolecules.education.fr/
. Le site de référence est http://www.rcsb.org/pdb/
ou une des sites miroir comme http://www.pdb.mdc-berlin.de/pdb/
. Mais il est préférable de se faire sa propre
base de données de molécules car les
recherches ne sont pas simples. La mienne se trouve dans le
répertoire jmol/PDB/.
Une excellente adresse de la simualisation
de l'ADN avec Jmol (en espagnol: http://www2.uah.es/biomodel/model4/dna/es/index.htm,
voir plus précisément la page http://www2.uah.es/biomodel/model4/dna/es/dnapairs.htm).
Le texte est excellent et les explications très
claires.
|
|
|
|
Remarques importantes:
les fichiers de structures sont des fichiers qui
décrivent tout simplement les coordonnées (0, x,
y, z) de chaque atome. La plupart des résultats ont
été obtenus par cristallographie sur des
composés purifiés et cristallisés. Mais
on notera que la cristallographie X ne permet pas de
positionner avec certitude les hydrogènes (sauf en
diffraction de neutrons). Les grosses molécules sont
donc données sans les atomes d'hydrogène,
notamment l'ADN ou les ARN. Pour les molécules de
petite taille les hydrogènes sont cependant parfois
placés de façon théorique.
|
|

|
|
Cliché de diffraction de l'ADN
obtenu par Rosalind
Franklin et Maurice Wilkins du
Collège King de Londres et publié en 1951. Il
permit de proposer la structure en double hélice de
l'ADNnote.
Les taches noires correspondent à des tâches de
diffraction qui reflètent la position ordonnée
des atomes qui composent la molécule. On notera que
de nombreuses protéines, notamment membranaires,
restent rebelles à toute cristallisation et donc
à une analyse par diffraction des RX (on peut par
contre utiliser d'autres méthodes spectroscopiques
mais plus délicates encore). Selon la
résolution du cliché on peut obtenir plus ou
moins d'information (0,5 nm de résolution est une
valeur assez faible qui ne permet d'obtenir qu'une carte
approximative de l'armature de la molécule alors que
0,15 nm est une résolution qui permet de positionner
avec précision tous les atomes sauf
l'hydrogène). L'analyse des clichés est
d'autant plus difficile que le nombre d'atomes est important
et que les symétries de la molécule sont peu
nombreuses.
La forme des molécules
simualisée ici en 3D n'est pas celle qu'elles peuvent
avoir dans une cellule vivante; leur forme dynamique in vivo
reste inaccessible
|
|
|
Représentation des
atomes (rayon de Van der Waals)
"ROTATION AUTOMATIQUE"
faire tourner lentement la molécule
autour d'un axe vertical (y)
Revenir à la position du début
Choisissez la
molécule à afficher
(bases, sucres,
groupement phosphate, nucléotides)
Je suis
désolé pour les utilisateurs de
Windows, je n'arrive pas à afficher les
codes corrects (Mac Roman) dans les scripts
(JmolMenu) pour les caractères
accentués; toute aide serait bienvenue.
|
|
codes couleurs
|
|
C
|
carbone
|
|
H
|
hydrogène
|
|
O
|
oxygène
|
|
N
|
azote
|
|
P
|
phosphore
|
|
|
Afficher ici deux molécules
simultanément pour les comparer
(bases, sucres,
groupement phosphate, nucléotides)
|
|
|
|
Les fonctions de la colonne de droite ne sont
que quelques fonctions destinées à vous
familiariser avec le logiciel.
|
|
- Pour accéder à toutes les
fonctions de l'applet, SOUS MAC cliquez sur le fond de
l'applet avec la touche CTRL enfoncée , SOUS WINDOWS
cliquez sur le fond de l'applet avec le BOUTON GAUCHE DE LA
SOURIS (il y a une aide dans les menus)
|
|
- Pour un ZOOM, SOUS MAC déplacez
votre souris (vers le haut ou le bas) EN MAINTENANT LA
TOUCHE ALT ENFONCÉE, sous WINDOWS, utilisez la
molette (l'applet étant sélectionnée
par un clic droit).
|
|
|
Molécule d'ADN
Double
hélice (forme B)
(fichier
pdb: 1BNA)
composée de 12 paires de
nucléotides de séquence
5'-CGCGAATTCGCG-3'
pour l'un des brins et
3'-GCGCTTAAGCGC-5'
pour l'autre brin.
Remarques:
- les H ne sont pas représentés
- la molécule d'ADN comporte 80
molécules d'H2O associées
(représentées par les atomes d'O).
représentation des atomes
(rayon de Van der Waals)
représentation des liaisons faibles entre
bases appariées (liaisons
hydrogène) (choisissez le menu
"calculer")
ROTATION AUTOMATIQUE
faire
tourner lentement la molécule autour d'un
axe vertical (y)
Revenir
à la position du début
|
codes couleurs
|
|
C
|
carbone
|
|
H
|
hydrogène
|
|
O
|
oxygène
|
|
N
|
azote
|
|
P
|
phosphore
|
|
|
|
|
|
Remarque: je précise que j'ai
bidouillé les fichiers .pdb originaux afin de
représenter ce qui m'intéresse
PÉDAGOGIQUEMENT. Le descriptif des atomes n'est plus
cohérent avec la molécule telle qu'elle a
été découverte et étudiée
à partir des clichés de diffraction. Pour une
étude scientifique se référer à
la base américaine de données (voir lien 1BNA).
|
|
les* ARN : acides ribonucléiques
|
|
|
|
* les ARN sont très divers en
forme, en taille et par leurs rôles.
Certains, très courts, de quelques
nucléotides à quelques dizaines de
nucléotides, ont des rôles très
récemment découverts comme
éléments interférant avec l'ADN (voir
aussi les travaux
de Beljanski). On
parle cependant de l'ARN comme un type de molécule
chimique.
|
|
Les* ARN sont une simple chaîne de nucléotides
(ce sont des molécules monocaténaires).
Les nucléotides d'une même chaîne sont
associés par des liaisons fortes.
|
|
La chaîne de nucléotides se replie facilement
sur elle-même et les bases complémentaires (A =U,
C = G) s'associent par des liaisons faibles.
|
|
Les nucléotides de l'ARN contiennent du ribose et 4
bases azotées (A, U, C, G).
|
|
nucléotide
(ribonucléotide)
|
|
|
nucléoside
(ribonucléoside)
|
+
|
groupement phosphate
|
|
|
base (azotée)
|
+
|
sucre
|
|
A
|
adénine
|
bases puriques

|
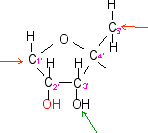
ribose
C5H10O5
|
PO43-
(en solution aqueuse, c'est
l'acide phosphorique: H3 PO4)
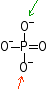
|
|
G
|
guanine
|
|
C
|
cytosine
|
bases pyrimidiques

|
|
U
|
uracile
|
|
la flèche
rouge indique
le point de liaison avec le sucre
du nucléotide auquel appartient la base
|
les flèches
rouges indiquent les
liaisons avec la base à gauche et le
groupement phosphate à droite
la flèche
verte indique le point
de liaison éventuel (en 3') avec le
groupement phosphate d'un autre
nucléotide
|
la flèche
rouge indique le point de
liaison avec le sucre (en 5') du
nucléotide auquel appartient le
groupement phosphate
la flèche
verte indique la liaison
éventuelle avec le sucre d'un autre
nucléotide
|
|
|
|
Simualiser les ARN avec un
logiciel 3D est beaucoup plus difficile car il existe un
très grand nombre de formes d'ARN différentes.
Nous réserverons cela à la classe de
1èreS.
|
|
note

|
|
L'histoire des sciences s'est efforcée
de rendre à Rosalind Franklin (photo)
la paternité de son travail (la structure de l'ADN
repose sur l'interprétation d'un cliché de
diffraction obtenu après des efforts innombrables et
persévérants) qui lui avait été
volé à la suite d'une présentation
informelle qu'en avait fait Wilkins à Jim
Watson le 6 février 1953 (fruit d'un dialogue
fécond entre chercheurs comme le rapporte Linus
Pauling, et aucunement de concurrence). Le
modèle de Watson et Crick fût
élaboré entre le 6 et le 28 février
à partir de ce cliché mais sans le citer.
D'une part parce que leur article de Nature était
TOTALEMENT SPÉCULATIF alors que justement le travail
des chimistes qui travaillaient sur les clichés de
diffraction consistait à rechercher patiemment des
PREUVES (l'article dénigrait même le travail
des expérimentateurs). Et d'autre part parce que
Watson ne voulait pas que l'on puisse le qualifier de
voleur. Ce qui ne manqua pas, malgré son prix Nobel.
Il s'en tira alors admirablement en publiant
un petit ouvrage de vulgarisation (The Double Helix)
dans lequel il raconta avec talent des fariboles sur une
prétendue course entre d'autres chercheurs et
lui-même. Il chargea la mémoire de Rosalind
Franklin (comme étant « une personne
asociale, dépourvue d''imagination, dogmatique et
obsédée par les détails de
l'expérience »), qui ne pouvait se
défendre, étant décédée.
|
|
Ces procédés malhonnêtes,
n'auraient aucun intérêt à être
relatés, s'ils n'éclairaient d'un jour nouveau
ce qui allait devenir une habitude en science : la fraude et
la course à la publication de résultats, afin
de s'assurer une position sociale, au détriment du
travail patient de l'expérimentateur.
|
|
La Raison malmenée.
De l'origine des idées reçues en biologie
moderne, Gérard Nissim Amzallag,
Préface d'André Pichot, CNRS
Editions 2002, ISBN :
978-2-271-05998-7, pour les
références historiques concernant cette
fraude voir pp137-140 (les faits rapportés ici
viennent de A. Sayre, Rosalind Franklin and DNA, 1975,
Norton and Company Inc, New York)
L'ouvrage de N. Amzallag est accessible
à un public cultivé non spécialiste;
après avoir fait un certain nombre de constats
justifiés historiquement, l'auteur propose une
refonte épistémologique de la recherche sur
le vivant: salutaire.
Je dirais
même indispensable si vous vous destinez à
une carrière de chercheur.
|